Ei pitäisi yllättää meitä, että jopa sellaisissa perimän osissa, joissa emme ilmeisesti näe ”funktionaalista” koodia (eli sellaista, joka on evolutiivisesti kiinnittynyt jonkin valikoivan edun seurauksena), on jonkinlaista koodia, mutta ei sellaista, jota olemme aiemmin pitäneet sellaisena. Entä jos se tekisi jotain kolmessa ulottuvuudessa ATGC-koodin yhden ulottuvuuden lisäksi? BioEssays-lehdessä juuri julkaistussa artikkelissa tutkitaan tätä kutkuttavaa mahdollisuutta…
Eikö olekin ihanaa, kun on todella hämmentävä ongelma, jota voi jyrsiä ja joka tuottaa lähes loputtomasti mahdollisia selityksiä. Entäpä ”mitä kaikkea ei-koodaava DNA tekee genomissa?” – se 98,5 % ihmisen geneettisestä materiaalista, joka ei tuota proteiineja. Oikeudenmukaisuuden nimissä voidaan todeta, että koodaamattoman DNA:n selvittäminen on edistynyt huomattavasti sellaisten sekvenssien tunnistamisen myötä, jotka transkriboituvat RNA:ksi, jotka muokkaavat geeniekspressiota, voivat siirtyä sukupolvelta toiselle (epigenetiikka) tai määrittää kantasolun tai tietyn kudossolun geeniekspressio-ohjelman. Monista genomeista on löydetty valtavia määriä toistosekvenssejä (muinaisten retrovirusten jäänteitä), jotka eivät taaskaan koodaa proteiineja, mutta ainakin on olemassa uskottavia malleja siitä, mitä ne tekevät evoluution kannalta (aina genomiparasiitista symbioosiin ja jopa isäntägenomin ”hyväksikäyttöön” geneettisen monimuotoisuuden tuottamiseksi, jonka varassa evoluutio toimii); Muuten, jotkut ei-koodaavat DNA:t tuottavat RNA:ta, joka hiljentää nämä retrovirussekvenssit, ja retrovirusten tunkeutumisen genomeihin uskotaan olleen valikoiva paine RNA-interferenssin (niin sanotun RNAi:n) evoluutiolle; toistuvia elementtejä eri nimityyppejä ja tandemtoistoja on runsaasti; intronit (joista monet sisältävät edellä mainittuja ei-koodaavia sekvenssityyppejä) ovat osoittautuneet ratkaisevan tärkeiksi geenien ilmentymisessä ja säätelyssä, mikä on huomattavinta, koska ne ovat erottaneet toisistaan vaihtoehtoisia koodaavia segmenttejä.
Siltikin on vielä paljon ongelmaa, jota pureskella, sillä vaikka ymmärrämme yhä paremmin suuren osan ei-koodaavan genomin luonnetta ja alkuperää ja olemme pääsemässä pitkälle sen ”toiminnassa” (joka määritellään tässä evolutiivisesti valittuna, edullistavana vaikutuksena isäntäorganismiin), emme ole vielä läheskään selittäneet kaikkea, ja – mikä tärkeintä – tarkastelemme sitä niin sanotusti hyvin pienellä linssillä. Yksi kiehtova asia DNA-sekvensseissä on se, että yksi sekvenssi voi ”koodata” useamman kuin yhden tiedon riippuen siitä, mikä sitä ”lukee” ja mihin suuntaan – virusgenomit ovat klassisia esimerkkejä siitä, että geenit, joita luetaan yhteen suuntaan tietyn proteiinin tuottamiseksi, menevät päällekkäin yhden tai useamman geenin kanssa, joita luetaan päinvastaiseen suuntaan (eli DNA:n komplementaarisesta säikeestä) eri proteiinien tuottamiseksi. Se on vähän kuin yksinkertaisten viestien tekeminen käänteisparin sanoilla (ns. emordnilap). Esim: REEDSTOPSFLOW, joka kuvitteellisella lukulaitteella voitaisiin jakaa muotoon REED STOPS FLOW. Takaperin luettuna se antaisi WOLF SPOTS DEER.
Nyt jos on evolutiivisesti edullista, että kaksi viestiä on koodattu niin taloudellisesti – kuten virusten genomeissa, joilla on taipumus kehittyä kohti minimaalista monimutkaisuutta informaatiosisällön suhteen, mikä vähentää lisääntymiseen tarvittavia resursseja – silloin viestit itsessään kehittyvät suurella rajoitteella. Mitä tämä tarkoittaa? No, voisimme muotoilla alkuperäisen esimerkkiviestimme muotoon RUSH-STEM IMPEDES CURRENT, joka sisältäisi saman olennaisen tiedon kuin REED STOPS FLOW. Tämä viesti ei kuitenkaan käänteisesti luettuna (tai jopa samassa merkityksessä, mutta eri osissa) koodaa mitään erityisen merkityksellistä. Todennäköisesti ainoa tapa välittää molemmat alkuperäisten viestien sisältämät tiedot samanaikaisesti on juuri sanamuoto REEDSTOPSFLOW: se on erittäin rajoitettu järjestelmä! Jos nimittäin tutkisimme riittävästi esimerkkejä englanninkielisistä käänteisparilauseista, huomaisimme, että ne koostuvat kaiken kaikkiaan melko lyhyistä sanoista, ja sekvensseistä puuttuvat tietyt kielen yksiköt, kuten artikkelit (the, a); jos katsoisimme tarkemmin, saattaisimme jopa havaita, että tällaisissa viesteissä tietyt aakkosten kirjaimet ovat keskimääräistä enemmän edustettuina. Näkisimme nämä vääristyminä sanojen ja kirjainten käytössä, jotka a priori antaisivat meille mahdollisuuden yrittää tunnistaa tällaisia ”kaksitoimisia” tiedonpalasia.
Palataanpa nyt takaisin genomiin koodattuihin ”kirjaimiin”, ”sanoihin” ja ”informaatioon”. Jos samaan geenisekvenssin osaan koodattaisiin kaksi erilaista informaation osaa, odottaisimme samalla tavalla, että rajoitteet ilmenisivät sanojen ja kirjainten käytön vinoutumina – analogiat vastaavasti proteiineja muodostavien aminohapposekvenssien ja niiden kolmen kirjaimen koodin osalta. Näin ollen DNA-sekvenssi voi koodata proteiinia ja lisäksi jotain muuta. Giorgio Bernardin mukaan tämä ”jokin muu” on tietoa, joka ohjaa solun valtavan pitkän DNA:n pakkaamista suhteellisen pieneen ytimeen. Kyseessä on ensisijaisesti koodi, joka ohjaa DNA:n pakkaamiseen käytettävien histoneiksi kutsuttujen proteiinien sitoutumista. Bernardi kutsuu tätä ”genomikoodiksi” – rakenteelliseksi koodiksi, joka määrittelee DNA:n muodon ja pakkautumisen ”kromatiiniksi” kutsuttuun erittäin tiivistyneeseen muotoon.
Mutta emmekö me aloittaneet selittämällä ei-koodaavaa DNA:ta, emmekä proteiineja koodaavia sekvenssejä? Kyllä, ja pitkissä ei-koodaavan DNA:n pätkissä näemme informaatiota, joka ylittää pelkkiä toistoja, tandemtoistoja ja muinaisten retrovirusten jäänteitä: on olemassa eräänlainen koodi tasolla, jossa kemiallisen DNA:n emästen GC-paria suositaan AT:hen verrattuna. Kuten Bernardi tarkastelee, syntetisoiden hänen ja muiden uraauurtavaa työtä, eukaryoottisen genomin ydinsekvensseissä GC-pitoisuus genomin rakenteellisissa organisatorisissa yksiköissä, joita kutsutaan ”isokuoriksi”, lisääntyi evolutiivisen siirtymän aikana niin sanottujen kylmäveristen ja lämminveristen organismien välillä. Ja kiehtovaa kyllä, tämä sekvenssivinouma osuu päällekkäin sellaisten sekvenssien kanssa, jotka ovat toiminnaltaan paljon rajoitetumpia: nämä ovat juuri edellä mainittuja proteiineja koodaavia sekvenssejä, ja ne – enemmän kuin välissä olevat ei-koodaavat sekvenssit – ovat johtolanka ”genomikoodista”.
Proteiineja koodaavat sekvenssit ovat myös pakattuina ja tiivistettyinä ytimessä – erityisesti silloin, kun ne eivät ole ”käytössä” (ts, transkriboidaan ja sitten käännetään proteiiniksi) – mutta ne sisältävät myös suhteellisen jatkuvaa tietoa tarkoista aminohappojen identiteeteistä, muuten ne eivät koodaisi proteiineja oikein: evoluutio toimisi tällaisiin mutaatioihin erittäin negatiivisella tavalla, mikä tekisi äärimmäisen epätodennäköiseksi, että ne säilyisivät ja näkyisivät meille. DNA:n aminohappokoodissa on kuitenkin pieni ”juju”, joka kehittyi yksinkertaisimmissa yksisoluisissa eliöissä (bakteereissa ja arkeoissa) miljardeja vuosia sitten: koodi on osittain tarpeeton. Esimerkiksi aminohappo treoniini voidaan koodata eukaryoottisessa DNA:ssa peräti neljällä eri tavalla: ACT, ACC, ACA tai ACG. Kolmas kirjain on muuttuva ja siten ”käytettävissä” ylimääräisen informaation koodaamiseen. Juuri näin tapahtuu ”genomikoodin” luomiseksi, ja tässä tapauksessa ACC- ja ACG-muotoja suositaan lämminverisissä eliöissä. Tämän ylimääräisen ”koodin” korkea rajoitus – joka on nähtävissä myös sellaisissa genomin osissa, joihin ei kohdistu tällaisia rajoituksia, kuten proteiineja koodaaviin sekvensseihin – johtuu siis sellaisten proteiineja koodaavien sekvenssien pakkautumisesta, jotka sisältävät kahta informaatiokokonaisuutta samanaikaisesti. Tämä on analoginen esimerkkiimme erittäin rajoitetusta kaksoisinformaatiosekvenssistä REEDSTOPSFLOW.
Tärkeää on kuitenkin se, että rajoitus ei ole yhtä tiukka kuin englanninkielisessä esimerkissämme, koska aminohappoja kuvaavan triplettikoodin kolmannen paikan koodaus on redundanttia: parempi analogia olisi SHE*ATE*STU*, jossa asteriski tarkoittaa muuttuvaa kirjainta, jolla ei ole mitään merkitystä koneelle, joka lukee nelikirjaimisen viestin kolmikirjaimisen osan. Voisi sitten kuvitella toisen tietotason, joka muodostuu lisäämällä ”D” näihin tähtipisteisiin, jolloin saadaan SHEDATEDSTUD (SHE DATED STUD). Seuraavaksi kuvitellaan toinen lukukone, joka etsii ”arkaluonteisia” merkityksellisiä lauseita, jotka sisältävät keskimääräistä enemmän D-kirjaimia. Tällä lukukoneella on mukanaan taittokone, joka asettaa eräänlaisen tapin jokaisen D:n kohdalle, jolloin viestiä taivutetaan 120 astetta tasossa. pisteessä, jossa viestiä pitäisi taivuttaa 120 astetta samassa tasossa, saisimme lopputulokseksi tiiviimmän, kolmiomaisen version. Eukaryoottisissa genomeissa GC-sekvenssivinouma, jonka on ehdotettu olevan vastuussa rakenteellisesta tiivistymisestä, ulottuu ei-koodaaviin sekvensseihin, joista joillakin on tunnistettuja aktiviteetteja, vaikkakin niiden sekvenssi on vähemmän rajoitettu kuin proteiineja koodaavan DNA:n. Siellä se ohjaa niiden tiivistymistä histonia sisältävien nukleosomien kautta kromatiiniksi.
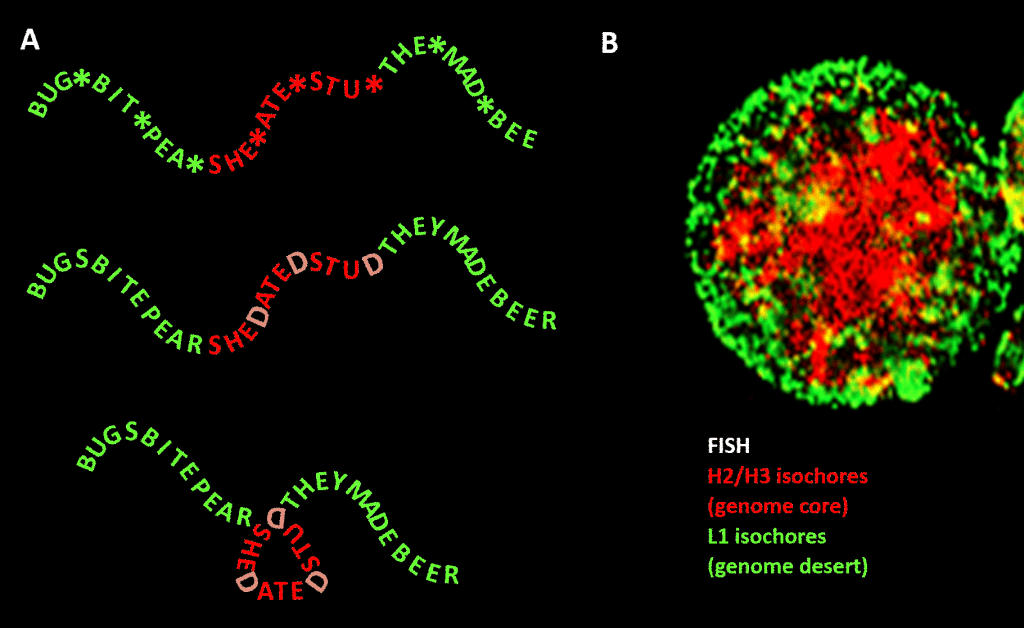
Kuva. Analogia sanapohjaisen viestin tiivistymisen ja genomisen DNA:n tiivistymisen välillä solun ytimessä. Paneeli A: Informaatio informaation sisällä, sanasarja, jossa on vaihteleva neljäs välilyönti, joka täytettynä tietyillä kirjaimilla synnyttää uuden viestin. Yhden viestin lukee kolmen kirjaimen lukukone, toisen lukukone, joka pystyy tulkitsemaan informaatiota, joka ulottuu sarjan neljänteen muuttuja-asemaan asti. Toinen lukukone tunnistaa ”arkaluonteisen” tiedon, joka pitäisi salata, ja niissä kohdissa, joissa 4. muuttujan kohdalla esiintyy D-kirjain, se taittaa sanajonon, jolloin ”arkaluonteinen” osa tiivistyy ja katoaa näkyvistä. Tämä on analogia kromatiinin kautta tapahtuvan genomisen 3D-pakkauksen periaatteelle, kuten paneelissa B on esitetty: fluoresenssikuva (fluoresenssi-in-situ-hybridisaation (FISH) avulla) solun ytimestä. H2/H3-isokuoret, joiden GC-pitoisuus kasvoi evoluution aikana kylmäverisistä lämminverisiksi selkärankaisiksi, ovat pakkautuneet kromatiiniytimeen, jolloin L1-isokuoret (joiden GC-pitoisuus on alhaisempi) jäävät periferiaan vähemmän pakkautuneeseen tilaan. Bernardin mukaan solun nukleosomien sijoittelukoneisto lukee genomin korkean GC-pitoisuuden omaaviin osiin sisältyvän ”genomikoodin” ja tulkitsee sen sekvenssiksi, joka on tiivistettävä voimakkaasti euchromatiiniin. Kiitokset: Paneeli A: konsepti ja kuvien tuottaminen: Paneeli B: H2/H3- ja L1-isokuorien FISH-kuvio PHA:n indusoimasta lymfosyytistä – S. Sacconen suosittelema – kuten jäljennetty artikkelissa Ref. .].
Tällöin näitä DNA:n alueita voidaan pitää rakenteellisesti tärkeinä elementteinä, jotka muodostavat oikean muodon ja erottelun genomissa tiivistetyille koodaaville sekvensseille riippumatta mistään muusta mahdollisesta funktiosta, joka näillä ei-koodaavilla sekvensseillä on: pohjimmiltaan tämä olisi ”selitys” sellaisten sekvenssien säilymiselle genomissa, joille ei voida osoittaa mitään ”funktiota” (evoluutiossa valikoituneen aktiivisuuden kannalta) (tai ainakaan merkittävää funktiota ei voida osoittaa).
Viimeinen analogia – tällä kertaa paljon läheisemmin liittyvä – voisi olla juuri aminohapposekvenssit suurissa proteiineissa, jotka tekevät erilaisia kierroksia, käännöksiä, poimuja jne. Saatamme ihmetellä tällaisia monimutkaisia rakenteita ja kysyä ”mutta tarvitseeko niiden olla aivan näin monimutkaisia, jotta ne toimisivat?”. Ehkä niiden on oltava monimutkaisia, jotta proteiinin osat voidaan tiivistää ja sijoittaa juuri siihen suuntaan ja paikkaan, joka tuottaa kolmiulotteisen rakenteen, jonka evoluutio on valinnut menestyksekkäästi. Mutta kun tiedämme, että ”genomikoodi” on päällekkäin proteiineja koodaavien sekvenssien kanssa, saatamme jopa alkaa epäillä, että toiminnassa on toinenkin valikoiva paine…
Andrew Moore, Ph.D.
Editor-in-Chief, BioEssays