Não nos deve surpreender que mesmo em partes do genoma onde obviamente não vemos um código “funcional” (ou seja, que tenha sido corrigido evolutivamente como resultado de alguma vantagem selectiva), exista um tipo de código, mas não como nada que tenhamos considerado anteriormente como tal. E se ele estivesse fazendo algo em três dimensões, assim como a única dimensão do código ATGC? Um artigo recém publicado em BioEssays explora essa possibilidade tentadora…
Não é maravilhoso ter um problema realmente perplexo para roer, um problema que gera explicações potenciais quase infinitas. Que tal “o que faz todo aquele DNA não codificador em genomas” – que 98,5% do material genético humano que não produz proteínas. Para ser justo, a decifração do DNA não codificador está dando grandes passos através da identificação de sequências que são transcritas em RNAs que modulam a expressão gênica, podem ser transmitidas transgeneracionalmente (epigenética) ou definir o programa de expressão gênica de uma célula tronco ou célula tecidual específica. Quantidades maciças de sequências repetidas (remanescentes de retrovírus antigos) foram encontradas em muitos genomas, e mais uma vez, estes não codificam as proteínas, mas pelo menos existem modelos credíveis para o que estão a fazer em termos evolutivos (desde o parasitismo genómico à simbiose e até à “exploração” pelo próprio genoma hospedeiro para produzir a diversidade genética sobre a qual a evolução funciona); A propósito, alguns DNA não codificadores fazem RNAs que silenciam estas sequências retrovirais, e acredita-se que a ingressão retroviral nos genomas tenha sido a pressão selectiva para a evolução da interferência do RNA (o chamado RNAi); abundam elementos repetitivos de vários tipos nomeados e repetições tandem; os introns (muitos dos quais contêm os tipos de sequências não codificadoras acima mencionados) têm-se revelado cruciais na expressão e regulação do gene, mais notoriamente através da emenda alternativa dos segmentos codificadores que eles separam.
Pouca, há muitos problemas a roer porque embora estejamos cada vez mais compreendendo a natureza e origem de grande parte do genoma não codificador e estejamos fazendo grandes incursões em sua “função” (definida aqui como evolutivamente selecionada, efeito vantajoso sobre o organismo hospedeiro), estamos longe de explicar tudo isso, e – mais ao ponto – estamos olhando para ele com uma lente de muito baixa amplificação, por assim dizer. Uma das coisas intrigantes sobre as sequências de ADN é que uma única sequência pode “codificar” mais do que uma peça de informação dependendo do que a está a “ler” e em que direcção – os genomas virais são exemplos clássicos em que genes lidos numa direcção para produzir uma determinada sobreposição de proteínas com um ou mais genes lidos na direcção oposta (ou seja, a partir da cadeia complementar do ADN) para produzir proteínas diferentes. É um pouco como fazer mensagens simples com palavras de pares inversos (um chamado emordnilap). Por exemplo, o que é um emordnilap? REEDSTOPSFLOW, que, por um dispositivo de leitura imaginário, poderia ser dividido em REED STOPS FLOW. Lido ao contrário, daria ao WOLF SPOTS DEER.
Agora, se for de vantagem evolutiva que duas mensagens sejam codificadas de forma tão económica – como é o caso dos genomas virais, que tendem a evoluir para uma complexidade mínima em termos de conteúdo de informação, reduzindo assim os recursos necessários para a reprodução – então as próprias mensagens evoluem com um elevado grau de restrição. O que é que isto significa? Bem, poderíamos formular nossa mensagem original de exemplo como RUSH-STEM IMPEDES CURRENT, que incorporaria a mesma informação essencial que REED STOPS FLOW. Entretanto, essa mensagem, se lida ao contrário (ou mesmo no mesmo sentido, mas em pedaços diferentes) não codifica nada adicional que seja particularmente significativo. Provavelmente a única forma de transmitir as duas informações nas mensagens originais simultaneamente é o próprio texto REEDSTOPSFLOW: esse é um sistema altamente limitado! Na verdade, se estudássemos exemplos suficientes de frases de par inverso em inglês, veríamos que elas são, no conjunto, compostas de palavras bastante curtas, e que faltam certas unidades de linguagem como artigos (o, a); se olhássemos mais de perto, poderíamos até detectar uma representação maior do que a média de certas letras do alfabeto em tais mensagens. Nós as veríamos como tendenciosidades no uso de palavras e letras que, a priori, nos permitiriam ter uma facada na identificação dessas informações de “dupla função”.
Agora vamos voltar às “letras”, “palavras”, e “informação” codificadas em genomas. Para que duas informações distintas sejam codificadas no mesmo pedaço de seqüência genética, esperaríamos, de forma semelhante, que as restrições se manifestassem em enviesamentos de uso de palavras e letras – as analogias, respectivamente, para as seqüências de aminoácidos que constituem proteínas, e seu código de três letras. Assim, uma sequência de DNA pode codificar para uma proteína e, além disso, para algo mais. Esta “outra coisa”, segundo Giorgio Bernardi, é informação que direciona a embalagem do enorme comprimento do DNA em uma célula para o núcleo relativamente minúsculo. Em primeiro lugar, é o código que orienta a ligação das proteínas de embalagem do DNA conhecidas como histones. Bernardi refere-se a isto como o “código genômico” – um código estrutural que define a forma e compactação do DNA na forma altamente condensada conhecida como “cromatina”.
Mas não começamos com uma explicação para DNA não codificador, não seqüências codificadoras de proteínas? Sim, e nos longos trechos de DNA não codificador vemos informações em excesso de meras repetições, repetições tandem e restos de retrovírus antigos: há um tipo de código no nível de preferência para o par de bases de DNA químico GC em comparação com AT. Como Bernardi revisa, sintetizando seu trabalho pioneiro e de outros, nas seqüências centrais do genoma eucariótico, o conteúdo do GC em unidades organizacionais estruturais do genoma denominado “isochores” aumentou durante a transição evolutiva entre os chamados organismos de sangue frio e de sangue quente. E, fascinantemente, esse viés de seqüência se sobrepõe a seqüências que são muito mais restritas em função: essas são as próprias seqüências codificadoras de proteínas mencionadas anteriormente, e elas – mais do que as seqüências não codificadoras intervenientes – são a pista para o “código genômico”.
Seqüências codificadoras de proteínas também são embaladas e condensadas no núcleo – particularmente quando elas não estão “em uso” (ou seja mas também contêm informação relativamente constante sobre identidades precisas de aminoácidos, caso contrário não codificariam as proteínas correctamente: a evolução actuaria sobre tais mutações de uma forma altamente negativa, tornando-as extremamente improváveis de persistir e serem visíveis para nós. Mas o código dos aminoácidos no DNA tem uma pequena “pegada” que evoluiu nos mais simples organismos unicelulares (bactérias e arquebactérias) bilhões de anos atrás: o código é parcialmente redundante. Por exemplo, o aminoácido Threonine pode ser codificado em ADN eucariótico de nada menos que quatro formas: ACT, ACC, ACA ou ACG. A terceira letra é variável e, portanto, “disponível” para a codificação de informação extra. Isto é exatamente o que acontece para produzir o “código genômico”, neste caso criando um viés para as formas ACC e ACG em organismos de sangue quente. Portanto, a alta restrição a este “código” adicional – que também é vista em partes do genoma que não estão sob tal restrição como seqüências codificadoras de proteínas – é imposta pela embalagem de seqüências codificadoras de proteínas que incorporam dois conjuntos de informações simultaneamente. Isto é análogo ao nosso exemplo da sequência de dupla informação altamente limitada REEDSTOPSFLOW.
Importante, no entanto, a restrição não é tão rigorosa como no nosso exemplo em inglês devido à redundância da terceira posição do código triplet para os aminoácidos: uma melhor analogia seria SHE*ATE*STU* onde o asterisco representa uma letra variável que não faz qualquer diferença para a máquina que lê o componente de três letras da mensagem de quatro letras. Poder-se-ia então imaginar um segundo nível de informação formado pela adição de “D” nestes pontos de asterisco, para fazer SHEDATEDSTUD (SHE DATED STUD). Em seguida, imagine uma segunda máquina de leitura que procura frases significativas de “natureza sensível” contendo uma concentração maior que a média de Ds. Esta máquina de leitura leva consigo uma máquina dobrável que coloca uma espécie de pino em cada D, dobrando a mensagem em 120 graus num plano. um ponto onde a mensagem deve ser dobrada em 120 graus no mesmo plano, acabaríamos por ter uma versão mais compacta, triangular. Em genomas eucarióticos, o viés de sequência GC proposto para ser responsável pela condensação estrutural estende-se a sequências não codificadoras, algumas das quais identificaram actividades, embora menos limitadas em sequência do que o ADN codificador de proteínas. Aí ele direciona sua condensação através de nucleossomos contendo história para formar cromatina.
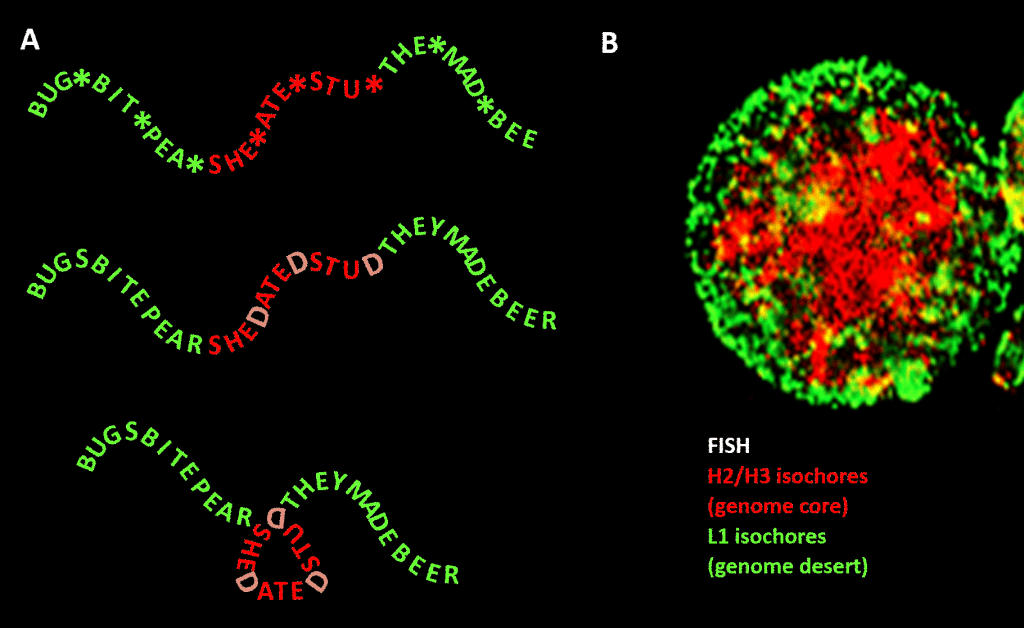
Figure. Analogia entre a condensação de uma mensagem baseada em palavras e a condensação de DNA genômico no núcleo da célula. Painel A: Informação dentro da informação, uma sequência de palavras com um quarto espaço variável que, quando preenchido com letras particulares, gera uma mensagem adicional. Uma mensagem é lida por uma máquina de leitura de três letras; a outra, por uma máquina de leitura que pode interpretar informação que se estende até à quarta posição variável da sequência. O segundo leitor reconhece a informação “sensível” que deve ser ocultada, e nos pontos onde aparece um “D” na 4ª posição, dobra a cadeia de palavras, comprimindo assim a parte “sensível” e retirando-a da vista. Esta é uma analogia para o princípio da compressão genómica 3D via cromatina, tal como descrito no painel B: uma imagem fluorescente (via Hibridização In-Situ Fluorescência – FISH) do núcleo celular. Os isócoros H2/H3, que aumentaram no conteúdo de GC durante a evolução de vertebrados de sangue frio para vertebrados de sangue quente, são comprimidos para um núcleo de cromatina, deixando os isócoros L1 (com menor conteúdo de GC) na periferia em um estado menos condensado. O “código genômico” incorporado nas vias de alta GC do genoma é, segundo Bernardi , lido pelas máquinas de posicionamento do nucleossoma da célula e interpretado como seqüência a ser altamente comprimida em eucromatina. Agradecimentos: Painel A: conceito e produção de figuras: Andrew Moore; Painel B: Um padrão FISH de isócoros H2/H3 e L1 a partir de um linfócito induzido por PHA – cortesia de S. Saccone – como reproduzido no Ref. .]
Estas regiões de DNA podem então ser consideradas como elementos estruturalmente importantes na formação da forma correta e separação das seqüências de codificação condensada no genoma, independentemente de qualquer outra função possível que essas seqüências não codificadoras tenham: em essência, isto seria uma “explicação” para a persistência em genomas de seqüências às quais nenhuma “função” (em termos de atividade evolucionária selecionada), pode ser atribuída (ou, pelo menos, nenhuma função substancial).
Uma analogia final – este tempo muito mais relacionado – pode ser as próprias sequências de aminoácidos em proteínas grandes, que fazem uma variedade de torções, voltas, dobras, etc. Podemos maravilhar-nos com estruturas tão complicadas e perguntar “mas será que elas precisam de ser tão complicadas para a sua função? Bem, talvez eles façam para condensar e posicionar partes da proteína na orientação e lugar exato que gera a estrutura tridimensional que foi selecionada com sucesso pela evolução. Mas com o conhecimento de que o “código genômico” se sobrepõe às seqüências de codificação protéica, podemos até começar a suspeitar que há outra pressão seletiva também no trabalho…
Andrew Moore, Ph.D.
Editor em Chefe, BioEssays