Nem lepődhetünk meg azon, hogy még a genom olyan részein is, ahol nyilvánvalóan nem látunk “funkcionális” kódot (vagyis olyat, amely evolúciósan rögzült valamilyen szelektív előny eredményeként), van egyfajta kód, de nem olyan, mint amit korábban annak tekintettünk. És mi lenne, ha az ATGC-kód egy dimenziója mellett három dimenzióban is csinálna valamit? A BioEssays-ben most megjelent tanulmány ezt a kínzó lehetőséget vizsgálja…
Hát nem csodálatos, ha van egy igazán zavarba ejtő probléma, amin rágódhatunk, ami szinte végtelen számú lehetséges magyarázatot generál. Mit szólnánk ahhoz, hogy “mit keres a genomban az a sok nem kódoló DNS?” – az emberi genetikai anyag 98,5%-a, amely nem termel fehérjéket. Az igazsághoz tartozik, hogy a nem kódoló DNS megfejtése nagy előrelépéseket tesz azon szekvenciák azonosítása révén, amelyek RNS-ekké íródnak át, amelyek módosítják a génexpressziót, transzgenerációsan továbbadhatók (epigenetika), vagy beállítják egy őssejt vagy egy adott szöveti sejt génexpressziós programját. Számos genomban találtak hatalmas mennyiségű ismétlődő szekvenciát (ősi retrovírusok maradványait), és ezek megint csak nem fehérjét kódolnak, de legalább vannak hiteles modellek arra, hogy evolúciós szempontból mit csinálnak (a genomiális parazitizmustól kezdve a szimbiózison át egészen a szimbiózisig, sőt a genetikai sokféleség előállításához, amelyen az evolúció működik, magának a gazdagenomnak a “kihasználásáig”); egyébként néhány nem kódoló DNS olyan RNS-t állít elő, amely elhallgattatja ezeket a retrovírus szekvenciákat, és a retrovírusok genomokba való behatolása feltehetően az RNS-interferencia (az úgynevezett RNAi) evolúciójának szelekciós nyomása volt; a különböző nevű repetitív elemek és tandem ismétlődések bővelkednek; az intronok (amelyek közül sok tartalmazza a fent említett típusú nem kódoló szekvenciákat) kulcsfontosságúnak bizonyultak a génexpresszió és a szabályozás szempontjából, leginkább az általuk elválasztott kódoló szegmensek alternatív splicingje révén.
Még mindig van min rágódni, mert bár egyre jobban megértjük a nem kódoló genom nagy részének természetét és eredetét, és jelentős eredményeket érünk el a “funkcióját” illetően (amit itt evolúciósan kiválasztott, a gazdaszervezetre gyakorolt előnyös hatásként definiálunk), még messze nem magyarázunk meg mindent, és – ami még fontosabb – úgymond nagyon kis nagyítású lencsével nézzük. A DNS-szekvenciák egyik érdekes tulajdonsága, hogy egyetlen szekvencia több információt is “kódolhat”, attól függően, hogy mi és milyen irányban “olvassa” azt – a vírusgenomok klasszikus példák arra, hogy az egyik irányban olvasott gének egy adott fehérje előállítása érdekében átfedésben vannak egy vagy több, az ellenkező irányban (azaz a DNS komplementer száláról) olvasott génnel, amelyek különböző fehérjéket állítanak elő. Kicsit olyan ez, mintha egyszerű üzeneteket készítenénk fordított párosítású szavakkal (úgynevezett emordnilap). Például: REEDSTOPSFLOW, amelyet egy képzeletbeli olvasóeszközzel REED STOPS FLOW-ra lehetne osztani. Visszafelé olvasva ez WOLF SPOTS DEER-t adna.
Most, ha evolúciós előnye van annak, hogy két üzenetet ilyen gazdaságosan kódoljanak – mint a vírusgenomokban, amelyek az információtartalom szempontjából a minimális komplexitás felé hajlamosak fejlődni, ezáltal csökkentve a szaporodáshoz szükséges erőforrásokat -, akkor maguk az üzenetek nagyfokú kényszerrel fejlődnek. Mit jelent ez? Nos, eredeti példaüzenetünket úgy is megfogalmazhatnánk, hogy RUSH-STEM IMPEDES CURRENT, ami ugyanazt a lényegi információt testesítené meg, mint a REED STOPS FLOW. Ez az üzenet azonban, ha fordítva olvassuk (vagy akár ugyanabban az értelemben, de más darabokban), nem kódol semmi olyan többletet, ami különösebb jelentőséggel bírna. Valószínűleg az egyetlen módja annak, hogy az eredeti üzenetekben szereplő mindkét információt egyszerre közvetítsük, maga a REEDSTOPSFLOW megfogalmazás: ez egy erősen korlátozott rendszer! Valóban, ha elég példát tanulmányoznánk az angol fordított páros mondatokra, azt látnánk, hogy ezek összességében meglehetősen rövid szavakból állnak, és a szekvenciákból hiányoznak bizonyos nyelvi egységek, például a cikkek (the, a); ha jobban megnéznénk, talán még az ábécé egyes betűinek átlagosnál nagyobb arányú képviseletét is felfedeznénk az ilyen üzenetekben. Ezeket a szó- és betűhasználat torzításainak tekintenénk, amelyek a priori lehetővé tennék számunkra, hogy megpróbáljuk azonosítani az ilyen “kettős funkciójú” információdarabokat.”
Most térjünk vissza a genomokban kódolt “betűkhöz”, “szavakhoz” és “információhoz”. Ha két különböző információt kódolnánk ugyanabban a genetikai szekvenciában, akkor hasonlóképpen elvárnánk, hogy a korlátok a szó- és betűhasználat torzításaiban nyilvánuljanak meg – a fehérjéket alkotó aminosav-szekvenciák, illetve azok hárombetűs kódjának analógiájára. Egy DNS-szekvencia tehát kódolhat egy fehérjét és emellett valami mást is. Ez a “valami más” Giorgio Bernardi szerint az az információ, amely a sejtben lévő hatalmas hosszúságú DNS-nek a viszonylag apró sejtmagba való becsomagolását irányítja. Elsősorban ez az a kód, amely a DNS-csomagoló fehérjék, a hisztonok kötődését irányítja. Bernardi ezt “genomi kódnak” nevezi – egy strukturális kódnak, amely meghatározza a DNS alakját és tömörítését a “kromatin” néven ismert, erősen tömörített formába.”
De nem a nem kódoló DNS magyarázatával kezdtük, nem a fehérjekódoló szekvenciákkal? Igen, és a nem kódoló DNS hosszú szakaszaiban a puszta ismétlődéseken, tandem ismétlődéseken és az ősi retrovírusok maradványain túlmutató információt látunk: van egyfajta kód a kémiai DNS-bázisok GC-párjának az AT-vel szembeni preferenciája szintjén. Ahogy Bernardi az ő és mások úttörő munkáját szintetizálva áttekinti, az eukarióta genom magszekvenciáiban az úgynevezett hidegvérű és melegvérű szervezetek közötti evolúciós átmenet során megnőtt a GC-tartalom a genom “izokóráknak” nevezett szerkezeti szervezeti egységeiben. És, ami lenyűgöző, ez a szekvencia-eltérés átfedésben van olyan szekvenciákkal, amelyek funkciójukat tekintve sokkal korlátozottabbak: ezek éppen a korábban említett fehérjekódoló szekvenciák, és ezek – jobban, mint a közbeeső nem kódoló szekvenciák – a “genomi kód” nyomai.
A fehérjekódoló szekvenciák is tömörülnek és tömörülnek a sejtmagban – különösen akkor, amikor nincsenek “használatban” (ill, átírják, majd fehérjévé fordítják) – de viszonylag állandó információt is tartalmaznak a pontos aminosav-azonosságokról, különben nem kódolnának helyesen fehérjéket: az evolúció rendkívül negatív módon hatna az ilyen mutációkra, ami rendkívül valószínűtlenné tenné, hogy fennmaradjanak és számunkra láthatóvá váljanak. A DNS-ben lévő aminosavkódnak azonban van egy kis “csapdája”, amely a legegyszerűbb egysejtű szervezetekben (baktériumok és archaea) fejlődött ki évmilliárdokkal ezelőtt: a kód részben redundáns. A treonin aminosav például nem kevesebb, mint négyféleképpen kódolható az eukarióta DNS-ben: ACT, ACC, ACA vagy ACG. A harmadik betű változó, és így “rendelkezésre áll” az extra információ kódolására. Pontosan ez történik a “genomikus kód” létrehozásakor, ami ebben az esetben a melegvérű szervezetekben az ACC és az ACG formák irányába mutat. Ezért ennek a kiegészítő “kódnak” a nagyfokú korlátozottságát – amely a genom olyan részein is megfigyelhető, amelyek nem állnak ilyen korlátozás alatt, mint a fehérjekódoló szekvenciák – a fehérjekódoló szekvenciák csomagolása okozza, amelyek egyszerre kétféle információt testesítenek meg. Ez analóg a mi példánkkal, a nagymértékben korlátozott, kettős információtartalmú REEDSTOPSFLOW szekvenciával.
Fontos azonban, hogy a korlátozás nem olyan szigorú, mint az angol nyelvű példánkban, mivel az aminosavakat jelölő triplet kód harmadik pozíciója redundáns: jobb analógia lenne a SHE*ATE*STU*, ahol a csillag egy változó betűt jelöl, amely nem tesz különbséget a négybetűs üzenet hárombetűs összetevőjét olvasó gép számára. Elképzelhető egy második információs szint, amely úgy jön létre, hogy ezekhez a csillagpontokhoz “D” betűt illesztünk, így jön létre a SHEDATEDSTUD (SHE DATED STUD). Ezután képzeljünk el egy második olvasógépet, amely olyan “érzékeny természetű” értelmes mondatokat keres, amelyek az átlagosnál nagyobb koncentrációban tartalmaznak D-ket. Ez az olvasógép egy hajtogatógépet hordoz magánál, amely minden egyes D-hez egyfajta csapot helyez, amely az üzenetet egy síkban 120 fokkal meghajlítja. egy olyan pontot, ahol az üzenetet ugyanabban a síkban 120 fokkal kellene meghajlítani, akkor egy tömörebb, háromszög alakú változatot kapnánk. Az eukarióta genomokban a szerkezeti sűrűsödésért felelősnek javasolt GC szekvencia torzítás kiterjed a nem kódoló szekvenciákra is, amelyek közül néhánynak azonosított aktivitása van, bár kevésbé korlátozott szekvenciájú, mint a fehérjekódoló DNS. Ott a hiszton-tartalmú nukleoszómákon keresztül irányítja ezek kondenzációját kromatin kialakítására.
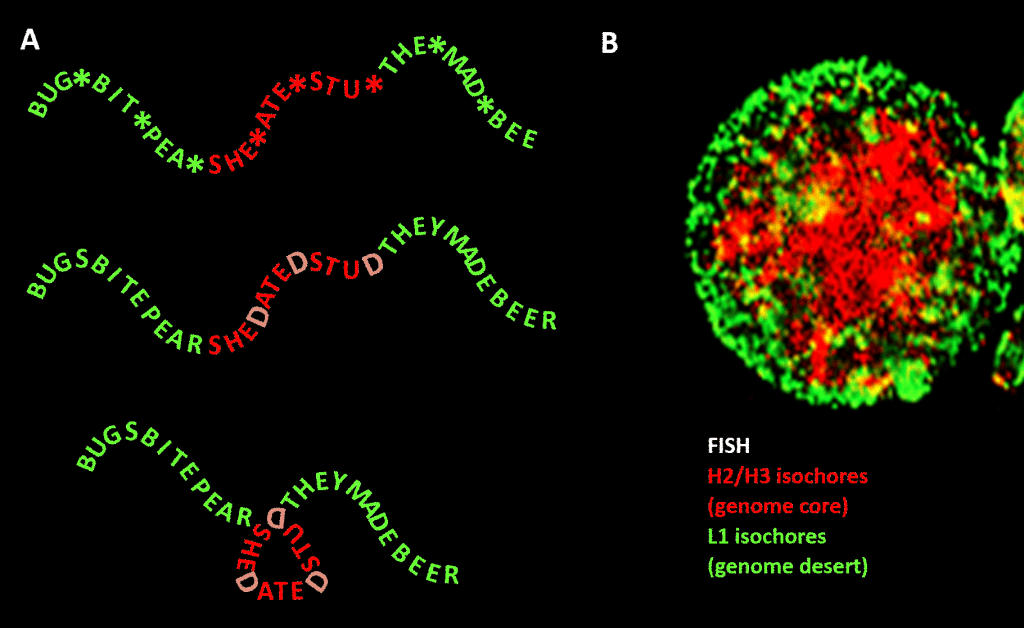
Ábra. Analógia egy szóalapú üzenet kondenzációja és a genomi DNS sejtmagban történő kondenzációja között. A panel: Információ az információban, szavak sorozata változó negyedik szóközzel, amelyet ha bizonyos betűkkel töltünk ki, egy további üzenetet hoz létre. Az egyik üzenetet egy hárombetűs olvasógép olvassa; a másikat egy olyan olvasógép, amely a szekvencia 4. változó-pozíciójáig terjedő információt képes értelmezni. A második olvasógép felismeri az “érzékeny” információkat, amelyeket el kell rejteni, és azokon a pontokon, ahol a 4. változós pozícióban egy “D” betű jelenik meg, összehajtja a szósorozatot, így az “érzékeny” részt összenyomja és eltünteti a szem elől. Ez a kromatinon keresztül történő genomi 3D tömörítés elvének analógiája, amint azt a B panel mutatja: a sejtmag fluoreszcens képe (fluoreszcens in situ hibridizációval – FISH). A H2/H3 izokórák, amelyek GC-tartalma nőtt a hidegvérűektől a melegvérű gerincesekig tartó evolúció során, egy kromatinmagba tömörülnek, és a periférián kevésbé tömörített állapotban hagyják az L1 izokórákat (alacsonyabb GC-tartalommal). A genom magas GC-tartalmú szakaszaiban megtestesülő “genomi kódot” Bernardi szerint a sejt nukleoszómapozícionáló gépezete olvassa, és szekvenciaként értelmezi, hogy az euchromatinban erősen tömörített legyen. Köszönetnyilvánítás: A panel: koncepció és az ábra készítése: B panel: H2/H3 és L1 izokórák FISH mintázata egy PHA által indukált limfocitából – S. Saccone jóvoltából – ahogyan a hivatkozásban is szerepel].
A DNS ezen régiói ezután szerkezetileg fontos elemeknek tekinthetők a genomban a kondenzált kódoló szekvenciák megfelelő alakjának és elkülönítésének kialakításában, függetlenül attól, hogy ezeknek a nem kódoló szekvenciáknak milyen más lehetséges funkciójuk van: lényegében ez lenne a “magyarázat” az olyan szekvenciák genomban való fennmaradására, amelyeknek nem tulajdonítható “funkció” (az evolúciósan kiválasztott aktivitás szempontjából) (vagy legalábbis nincs lényeges funkciójuk).
Egy utolsó analógia – ezúttal sokkal szorosabban kapcsolódó – a nagy fehérjékben található aminosav-szekvenciák lehetnek, amelyek különféle csavarokat, fordulatokat, hajtásokat stb. végeznek. Megcsodálhatjuk az ilyen bonyolult szerkezeteket, és megkérdezhetjük, hogy “de vajon szükségük van-e ilyen bonyolult szerkezetekre a funkciójukhoz?”. Nos, talán azért, hogy a fehérje részei pontosan abba az orientációba és helyre tömörüljenek és helyezkedjenek el, amely az evolúció által sikeresen kiválasztott háromdimenziós szerkezetet hozza létre. De annak ismeretében, hogy a “genomi kód” átfedésben van a fehérjéket kódoló szekvenciákkal, még az is gyanússá válhat, hogy egy másik szelekciós nyomás is működik…
Andrew Moore, Ph.D.
Editor-in-Chief, BioEssays