Det bør ikke overraske os, at selv i dele af genomet, hvor vi ikke tydeligvis kan se en “funktionel” kode (dvs. en kode, der er blevet evolutionært fastsat som følge af en eller anden selektiv fordel), er der en type kode, men ikke som noget, vi tidligere har betragtet som sådan. Og hvad nu hvis den gjorde noget i tre dimensioner ud over den ene dimension i ATGC-koden? En artikel, der netop er offentliggjort i BioEssays, udforsker denne fristende mulighed …
Er det ikke vidunderligt at have et virkelig forvirrende problem at gnave i, et problem, der genererer næsten uendelige potentielle forklaringer. Hvad med “hvad laver alt det ikke-kodende DNA i genomerne?” – de 98,5 % af det menneskelige genetiske materiale, som ikke producerer proteiner. Retfærdigvis skal det siges, at der gøres store fremskridt med at afkode ikke-kodende DNA ved at identificere sekvenser, der transskriberes til RNA’er, som modulerer genekspressionen, kan videregives transgenerationelt (epigenetik) eller fastlægger genekspressionsprogrammet for en stamcelle eller en specifik vævscelle. Der er fundet enorme mængder af gentagelsessekvenser (rester af gamle retrovirus) i mange genomer, og igen koder disse ikke for protein, men i det mindste findes der troværdige modeller for, hvad de gør i evolutionær henseende (lige fra genomisk parasitisme til symbiose og endog “udnyttelse” af selve værtsgenomet til at producere den genetiske diversitet, som evolutionen arbejder på); I øvrigt fremstiller noget ikke-kodende DNA RNA’er, der lukker munden på disse retrovirale sekvenser, og retroviral indtrængen i genomer menes at have været det selektive pres for udviklingen af RNA-interferens (såkaldt RNAi); gentagelseselementer af forskellige navngivne typer og tandemrepeats findes i overflod; introner (hvoraf mange indeholder de førnævnte typer af ikke-kodende sekvenser) har vist sig at være afgørende for genekspression og -regulering, især via alternativ splejsning af de kodningssegmenter, som de adskiller.
Der er stadig masser af problemer at gnave i, for selv om vi i stigende grad forstår arten og oprindelsen af en stor del af det ikke-kodende genom og gør store fremskridt med hensyn til dets “funktion” (her defineret som evolutionært udvalgte, fordelagtige virkninger på værtsorganismen), er vi langt fra at forklare det hele, og – endnu vigtigere – vi ser på det med en meget lav forstørrelseslinse, så at sige. En af de fascinerende ting ved DNA-sekvenser er, at en enkelt sekvens kan “kode” for mere end én information, afhængigt af hvad der “læser” den og i hvilken retning – virale genomer er klassiske eksempler, hvor gener, der læses i én retning for at producere et givet protein, overlapper med et eller flere gener, der læses i den modsatte retning (dvs. fra den komplementære DNA-streng) for at producere forskellige proteiner. Det er lidt ligesom at lave simple meddelelser med omvendt parrede ord (en såkaldt emordnilap). F.eks: REEDSTOPSFLOW, som ved hjælp af en imaginær læseanordning kunne opdeles i REED STOPS FLOW. Læses det baglæns, ville det give WOLF SPOTS DEER.
Nu, hvis det er en evolutionær fordel for to budskaber at være kodet så økonomisk – som det er tilfældet i virale genomer, der har en tendens til at udvikle sig i retning af minimal kompleksitet med hensyn til informationsindhold, hvorved de nødvendige ressourcer til reproduktion reduceres – så udvikler selve budskaberne sig med en høj grad af begrænsning. Hvad betyder dette? Vi kunne formulere vores oprindelige eksempelmeddelelse som RUSH-STEM IMPEDES CURRENT, hvilket ville indeholde den samme væsentlige information som REED STOPS FLOW. Men denne meddelelse, hvis den læses omvendt (eller endda i samme betydning, men i forskellige dele), koder ikke noget yderligere, der er særlig meningsfuldt. Den eneste måde, hvorpå begge oplysninger i de oprindelige meddelelser kan formidles samtidigt, er sandsynligvis selve ordlyden REEDSTOPSFLOW: det er et meget begrænset system! Hvis vi studerede tilstrækkeligt mange eksempler på omvendte sætninger på engelsk, ville vi faktisk se, at de i det store og hele består af ret korte ord, og at sekvenserne mangler visse sproglige enheder såsom artikler (the, a); hvis vi så nærmere efter, kunne vi endda opdage en større repræsentation end gennemsnittet af visse bogstaver i alfabetet i sådanne meddelelser. Vi ville se dette som skævheder i brugen af ord og bogstaver, som a priori ville give os mulighed for at få et forsøg på at identificere sådanne “dobbeltfunktionelle” stykker information.
Lad os nu vende tilbage til de “bogstaver”, “ord” og “information”, der er kodet i genomerne. Hvis to forskellige stykker information skulle være kodet i det samme stykke genetisk sekvens, ville vi på samme måde forvente, at begrænsningerne ville manifestere sig i en skævhed i brugen af ord og bogstaver – analogierne for henholdsvis aminosyresekvenser, der udgør proteiner, og deres trebogstavskode. En DNA-sekvens kan således både kode for et protein og for noget andet. Dette “andet” er ifølge Giorgio Bernardi den information, der styrer indpakningen af den enorme længde DNA i en celle i den relativt lille kerne. Det er først og fremmest koden, der styrer bindingen af de DNA-pakningsproteiner, der er kendt som histoner. Bernardi kalder dette for den “genomiske kode” – en strukturel kode, der definerer formen og komprimeringen af DNA’et i den stærkt kondenserede form, der er kendt som “kromatin”.
Men startede vi ikke med en forklaring på ikke-kodende DNA og ikke på protein-kodende sekvenser? Jo, og i de lange strækninger af ikke-kodende DNA ser vi information, der går ud over simple gentagelser, tandemrepeats og rester af gamle retrovirusser: Der er en type kode på niveauet af præference for GC-parret af kemiske DNA-baser i forhold til AT. Som Bernardi gennemgår i en sammenfatning af hans og andres banebrydende arbejde, er GC-indholdet i de centrale sekvenser af det eukaryote genom i de strukturelle organisatoriske enheder af genomet, kaldet “isokorer”, steget i løbet af den evolutionære overgang mellem såkaldte koldblodede og varmblodede organismer. Og fascinerende nok overlapper denne sekvensbias med sekvenser, der er meget mere begrænsede i deres funktion: det er netop de tidligere nævnte protein-kodende sekvenser, og de – mere end de mellemliggende ikke-kodende sekvenser – er ledetråden til den “genomiske kode”.
Protein-kodende sekvenser er også pakket og kondenseret i kernen – især når de ikke er “i brug” (dvs, ved at blive transskriberet og derefter oversat til protein) – men de indeholder også relativt konstante oplysninger om præcise aminosyreidentiteter, ellers ville de ikke kunne kode proteiner korrekt: evolutionen ville handle på sådanne mutationer på en meget negativ måde, hvilket ville gøre det yderst usandsynligt, at de ville bestå og være synlige for os. Men aminosyrekoden i DNA har en lille “hage”, som udviklede sig i de mest simple encellede organismer (bakterier og arkæer) for milliarder af år siden: koden er delvis redundant. F.eks. kan aminosyren Threonin kodes i eukaryotisk DNA på ikke færre end fire måder: ACT, ACC, ACA eller ACG. Det tredje bogstav er variabelt og dermed “tilgængeligt” for kodning af ekstra information. Det er præcis det, der sker for at frembringe den “genomiske kode”, hvilket i dette tilfælde skaber en skævhed til fordel for ACC- og ACG-formerne i varmblodede organismer. Den høje begrænsning af denne ekstra “kode” – som også ses i dele af genomet, der ikke er under en sådan begrænsning som proteinkodende sekvenser – pålægges derfor af pakningen af proteinkodende sekvenser, der indeholder to sæt informationer på samme tid. Dette er analogt med vores eksempel på den stærkt begrænsede sekvens med dobbelt information REEDSTOPSFLOW.
Væsentligt er det dog, at begrænsningen ikke er så streng som i vores eksempel på det engelske sprog på grund af redundansen i den tredje position i tripletkoden for aminosyrer: en bedre analogi ville være SHE*ATE*STU*, hvor stjernen står for et variabelt bogstav, der ikke gør nogen forskel for den maskine, der læser den tre bogstaver store del af den fire bogstaver store besked. Man kunne så forestille sig et andet informationsniveau, som dannes ved at tilføje et “D” ved disse stjernetegn, så man får SHEDATEDSTUD (SHE DATED STUD). Forestil dig dernæst en anden aflæsningsmaskine, der leder efter meningsfulde sætninger af “følsom karakter”, som indeholder en større koncentration af D’er end gennemsnittet. Denne læsermaskine har en foldemaskine med sig, der placerer en slags pløk ved hvert D og knækker budskabet 120 grader i et plan. et punkt, hvor budskabet skal bøjes 120 grader i samme plan, ville vi ende med en mere kompakt, trekantet version. I eukaryote genomer strækker den GC-sekvensbias, der foreslås at være ansvarlig for strukturel kondensering, sig ind i ikke-kodende sekvenser, hvoraf nogle har identificerede aktiviteter, selv om de er mindre begrænsede i sekvens end det protein-kodende DNA. Der styrer den deres kondensering via histonholdige nukleosomer for at danne kromatin.
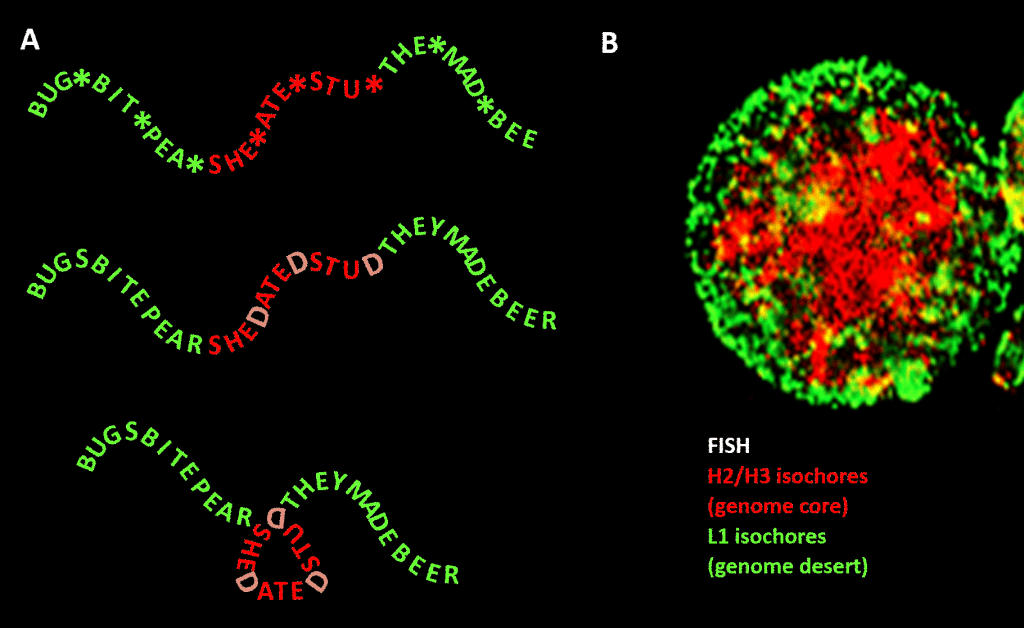
Figur. Analogi mellem kondensering af et ordbaseret budskab og kondensering af genomisk DNA i cellekernen. Panel A: Information i information, en sekvens af ord med et variabelt fjerde mellemrum, der, når det udfyldes med bestemte bogstaver, genererer en yderligere meddelelse. Det ene budskab læses af en læsemaskine med tre bogstaver; det andet af en læsemaskine, der kan fortolke information, der strækker sig til den fjerde variable position i sekvensen. Den anden læsemaskine genkender “følsomme” oplysninger, som bør skjules, og på de steder, hvor der forekommer et “D” i den fjerde position, folder den ordrækken, hvorved den komprimerer den “følsomme” del og fjerner den fra synligheden. Dette er en analogi til princippet om genomisk 3D-komprimering via kromatin, som vist i panel B: et fluorescensbillede (via Fluorescence In-Situ Hybridization – FISH) af cellekernen. H2/H3-isokorerne, hvis GC-indhold er steget i løbet af evolutionen fra koldblodede til varmblodede hvirveldyr, er komprimeret til en kromatinkerne, mens L1-isokorerne (med lavere GC-indhold) i periferien efterlades i en mindre komprimeret tilstand. Den “genomiske kode”, der er indeholdt i genomets høj-GC-strækninger, læses ifølge Bernardi af cellens nukleosom-placeringsmaskineri og fortolkes som en sekvens, der skal være stærkt komprimeret i euchromatin. Anerkendelser: Panel A: koncept og produktion af figurer: Andrew Moore; Panel B: Et FISH-mønster af H2/H3- og L1-isokore fra en lymfocyt induceret af PHA – med tak fra S. Saccone – som gengivet i Ref.]
Disse DNA-regioner kan så betragtes som strukturelt vigtige elementer til at danne den korrekte form og adskillelse af kondenserede kodningssekvenser i genomet, uafhængigt af enhver anden mulig funktion, som disse ikke-kodningssekvenser har: i det væsentlige ville dette være en “forklaring” på, at der i genomerne findes sekvenser, som ikke kan tilskrives nogen “funktion” (i form af evolutionært udvalgt aktivitet) (eller i det mindste ingen væsentlig funktion).
En sidste analogi – denne gang meget mere nært beslægtet – kunne være selve aminosyresekvenserne i store proteiner, som laver en række vridninger, drejninger, foldninger osv. Vi kan forundres over sådanne komplicerede strukturer og spørge “men behøver de at være helt så komplicerede for deres funktion?”. Tja, måske gør de det for at kondensere og placere dele af proteinet i den nøjagtige retning og på det nøjagtige sted, der genererer den tredimensionelle struktur, som evolutionen med held har udvalgt. Men med en viden om, at den “genomiske kode” overlapper de proteinkodende sekvenser, kan vi måske endda begynde at få mistanke om, at der også er et andet selektivt pres på spil…
Andrew Moore, Ph.D.
Hovedredaktør, BioEssays