No debería sorprendernos que incluso en partes del genoma donde no vemos obviamente un código «funcional» (es decir, uno que ha sido fijado evolutivamente como resultado de alguna ventaja selectiva), haya un tipo de código, pero no como algo que hayamos considerado previamente como tal. ¿Y si hiciera algo en tres dimensiones además de la dimensión única del código ATGC? Un artículo que acaba de publicarse en BioEssays explora esta tentadora posibilidad…
¿No es maravilloso tener un problema realmente desconcertante que roer, uno que genera explicaciones potenciales casi infinitas? Qué tal «¿qué hace todo ese ADN no codificante en los genomas?», ese 98,5% del material genético humano que no produce proteínas. Para ser justos, el desciframiento del ADN no codificante está avanzando a pasos agigantados gracias a la identificación de secuencias que se transcriben en ARN que modulan la expresión de los genes, que pueden transmitirse de forma transgeneracional (epigenética) o que establecen el programa de expresión genética de una célula madre o de un tejido específico. Se han encontrado cantidades masivas de secuencias repetidas (restos de antiguos retrovirus) en muchos genomas, y de nuevo, éstas no codifican para proteínas, pero al menos hay modelos creíbles de lo que hacen en términos evolutivos (que van desde el parasitismo genómico hasta la simbiosis e incluso la «explotación» por el propio genoma del huésped para producir la diversidad genética sobre la que trabaja la evolución); por cierto, algunos ADN no codificantes producen ARN que silencian estas secuencias retrovirales, y se cree que la entrada de retrovirus en los genomas ha sido la presión selectiva para la evolución del ARN de interferencia (el llamado ARNi); abundan los elementos repetitivos de varios tipos nombrados y las repeticiones en tándem; los intrones (muchos de los cuales contienen los tipos de secuencias no codificantes antes mencionados) han resultado ser cruciales en la expresión y regulación de los genes, sobre todo a través del empalme alternativo de los segmentos codificantes que separan.
Aún así, hay muchos problemas que roer porque, aunque cada vez entendemos mejor la naturaleza y el origen de gran parte del genoma no codificante y estamos haciendo grandes avances en su «función» (definida aquí como efecto ventajoso seleccionado evolutivamente en el organismo anfitrión), estamos lejos de explicarlo todo, y -más aún- lo estamos mirando con una lente de muy baja amplitud, por así decirlo. Uno de los aspectos más interesantes de las secuencias de ADN es que una misma secuencia puede «codificar» más de una información, dependiendo de qué la «lea» y en qué dirección: los genomas víricos son ejemplos clásicos en los que los genes que se leen en una dirección para producir una proteína determinada se solapan con uno o más genes que se leen en la dirección opuesta (es decir, desde la cadena complementaria del ADN) para producir proteínas diferentes. Es un poco como hacer mensajes simples con palabras de par inverso (el llamado emordnilap). Por ejemplo: REEDSTOPSFLOW, que, mediante un dispositivo de lectura imaginario, podría dividirse en REED STOPS FLOW. Leído al revés, daría WOLF SPOTS DEER.
Ahora bien, si resulta ventajoso desde el punto de vista evolutivo que dos mensajes estén codificados de forma tan económica -como es el caso de los genomas virales, que tienden a evolucionar hacia una complejidad mínima en términos de contenido informativo, reduciendo así los recursos necesarios para su reproducción-, entonces los propios mensajes evolucionan con un alto grado de restricción. ¿Qué significa esto? Bien, podríamos redactar nuestro mensaje de ejemplo original como RUSH-STEM IMPEDES CURRENT, lo que incorporaría la misma información esencial que REED STOPS FLOW. Sin embargo, ese mensaje, si se lee al revés (o incluso en el mismo sentido, pero en trozos diferentes) no codifica nada adicional que sea particularmente significativo. Probablemente, la única forma de transmitir simultáneamente las dos informaciones de los mensajes originales es la propia formulación REEDSTOPSFLOW: ¡es un sistema muy restringido! De hecho, si estudiáramos suficientes ejemplos de frases de par inverso en inglés, veríamos que, en general, están formadas por palabras más bien cortas, y en las secuencias faltan ciertas unidades del lenguaje, como los artículos (the, a); si miráramos más de cerca, podríamos incluso detectar una representación mayor que la media de ciertas letras del alfabeto en tales mensajes. Veríamos que se trata de sesgos en el uso de las palabras y las letras que, a priori, nos permitirían intentar identificar esas piezas de información de «doble función».
Volvamos ahora a las «letras», las «palabras» y la «información» codificada en los genomas. Para que dos piezas distintas de información estén codificadas en el mismo trozo de secuencia genética, esperaríamos, del mismo modo, que las restricciones se manifestaran en sesgos de uso de palabras y letras -las analogías, respectivamente, para las secuencias de aminoácidos que constituyen las proteínas, y su código de tres letras. Por tanto, una secuencia de ADN puede codificar una proteína y, además, otra cosa. Este «algo más», según Giorgio Bernardi, es la información que dirige el empaquetamiento de la enorme longitud de ADN de una célula en el relativamente pequeño núcleo. En primer lugar, es el código que guía la unión de las proteínas que empaquetan el ADN, conocidas como histonas. Bernardi se refiere a esto como el «código genómico», un código estructural que define la forma y la compactación del ADN en la forma altamente condensada conocida como «cromatina».
¿Pero no empezamos con una explicación para el ADN no codificante, no para las secuencias codificantes de proteínas? Sí, y en los largos tramos de ADN no codificante vemos información que va más allá de las meras repeticiones, de las repeticiones en tándem y de los restos de antiguos retrovirus: hay un tipo de código a nivel de preferencia por el par GC de las bases químicas del ADN frente al AT. Como repasa Bernardi, sintetizando su trabajo pionero y el de otros, en las secuencias centrales del genoma eucariota, el contenido de GC en las unidades organizativas estructurales del genoma denominadas «isócoras» aumentó durante la transición evolutiva entre los llamados organismos de sangre fría y los de sangre caliente. Y, fascinantemente, este sesgo de la secuencia se solapa con secuencias que están mucho más restringidas en su función: estas son las mismas secuencias codificadoras de proteínas mencionadas anteriormente, y ellas -más que las secuencias no codificadoras intermedias- son la pista del «código genómico».
Las secuencias codificadoras de proteínas también están empaquetadas y condensadas en el núcleo – particularmente cuando no están «en uso» (es decir, siendo transcritas, y luego traducidas a proteína) – pero también contienen información relativamente constante sobre las identidades precisas de los aminoácidos, de lo contrario fallarían en codificar correctamente las proteínas: la evolución actuaría sobre tales mutaciones de una manera altamente negativa, haciéndolas extremadamente improbables de persistir y ser visibles para nosotros. Pero el código de aminoácidos del ADN tiene una pequeña «trampa» que evolucionó en los organismos unicelulares más simples (bacterias y arqueas) hace miles de millones de años: el código es parcialmente redundante. Por ejemplo, el aminoácido treonina puede codificarse en el ADN eucariota nada menos que de cuatro maneras: ACT, ACC, ACA o ACG. La tercera letra es variable y, por tanto, «disponible» para la codificación de información adicional. Esto es exactamente lo que ocurre para producir el «código genómico», en este caso creando un sesgo para las formas ACC y ACG en los organismos de sangre caliente. Por lo tanto, la gran limitación de este «código» adicional -que también se observa en partes del genoma que no están sometidas a tal limitación como las secuencias codificadoras de proteínas- viene impuesta por el empaquetamiento de las secuencias codificadoras de proteínas que encarnan dos conjuntos de información simultáneamente. Esto es análogo a nuestro ejemplo de la secuencia de información dual altamente restringida REEDSTOPSFLOW.
Sin embargo, la restricción no es tan estricta como en nuestro ejemplo de la lengua inglesa debido a la redundancia de la tercera posición del código de tripletes para los aminoácidos: una mejor analogía sería SHE*ATE*STU* donde el asterisco representa una letra variable que no hace ninguna diferencia para la máquina que lee el componente de tres letras del mensaje de cuatro letras. A continuación, se podría imaginar un segundo nivel de información formado por la adición de «D» en estos puntos de asterisco, para hacer SHEDATEDSTUD (SHE DATED STUD). A continuación, imaginemos una segunda máquina lectora que busque frases significativas de «naturaleza sensible» que contengan una concentración de Ds superior a la media. Esta máquina lectora lleva consigo una máquina de plegado que coloca una especie de clavija en cada D, doblando el mensaje 120 grados en un plano. un punto en el que el mensaje debería doblarse 120 grados en el mismo plano, terminaríamos con una versión más compacta, triangular. En los genomas eucariotas, el sesgo de la secuencia GC propuesto como responsable de la condensación estructural se extiende a las secuencias no codificantes, algunas de las cuales tienen actividades identificadas, aunque menos constreñidas en su secuencia que el ADN codificador de proteínas. Allí dirige su condensación a través de nucleosomas que contienen histonas para formar la cromatina.
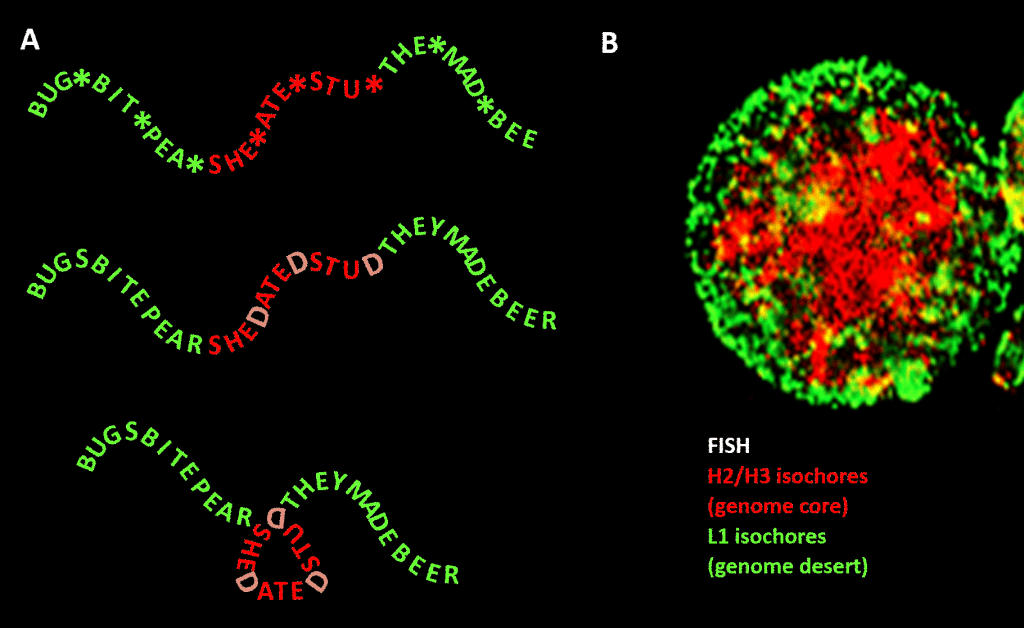
Figura. Analogía entre la condensación de un mensaje basado en palabras y la condensación del ADN genómico en el núcleo celular. Panel A: La información dentro de la información, una secuencia de palabras con un cuarto espacio variable que, al llenarse con determinadas letras, genera otro mensaje. Un mensaje es leído por una máquina lectora de tres letras; el otro por una máquina lectora que puede interpretar la información que se extiende hasta la cuarta posición variable de la secuencia. La segunda lectora reconoce la información «sensible» que debe ser ocultada, y en los puntos donde aparece una «D» en la 4ª posición, dobla la cadena de palabras, comprimiendo así la parte «sensible» y sacándola de la vista. Se trata de una analogía del principio de compresión tridimensional del genoma a través de la cromatina, como se muestra en el panel B: una imagen de fluorescencia (a través de la hibridación in situ de fluorescencia – FISH) del núcleo celular. Los isócoras H2/H3, que aumentaron su contenido de GC durante la evolución de los vertebrados de sangre fría a los de sangre caliente, se comprimen en un núcleo de cromatina, dejando los isócoras L1 (con menor contenido de GC) en la periferia en un estado menos condensado. El «código genómico» plasmado en los tramos de alto GC del genoma es, según Bernardi , leído por la maquinaria de posicionamiento de nucleosomas de la célula e interpretado como una secuencia que debe ser altamente comprimida en la eucromatina. Agradecimientos: Panel A: concepto y elaboración de la figura: Andrew Moore; Panel B: Un patrón FISH de isócoras H2/H3 y L1 de un linfocito inducido por PHA-cortesía de S. Saccone-como se reproduce en la Ref. .]
Estas regiones de ADN pueden considerarse entonces como elementos estructuralmente importantes en la formación de la forma correcta y la separación de las secuencias codificantes condensadas en el genoma, independientemente de cualquier otra función posible que tengan esas secuencias no codificantes: en esencia, esto sería una «explicación» para la persistencia en los genomas de secuencias a las que no se puede atribuir ninguna «función» (en términos de actividad seleccionada evolutivamente) (o, al menos, ninguna función sustancial).
Una analogía final-esta vez mucho más estrechamente relacionada-podría ser las propias secuencias de aminoácidos en las proteínas grandes, que hacen una variedad de giros, vueltas, pliegues etc. Podemos maravillarnos ante estructuras tan complicadas y preguntarnos «¿pero es necesario que sean tan complicadas para su función?». Bueno, tal vez sí para condensar y colocar partes de la proteína en la orientación y el lugar exactos que generan la estructura tridimensional que ha sido seleccionada con éxito por la evolución. Pero al saber que el «código genómico» se superpone a las secuencias codificantes de las proteínas, podríamos empezar a sospechar que también existe otra presión selectiva…
Andrew Moore, Ph.D.
Editor Jefe, BioEssays