Nu ar trebui să ne surprindă faptul că, chiar și în părțile genomului în care nu vedem în mod evident un cod „funcțional” (adică unul care a fost fixat evolutiv ca urmare a unui avantaj selectiv), există un tip de cod, dar nu ca tot ceea ce am considerat anterior ca atare. Și ce s-ar întâmpla dacă ar face ceva în trei dimensiuni, precum și în cea cu o singură dimensiune a codului ATGC? O lucrare care tocmai a fost publicată în BioEssays explorează această posibilitate ispititoare…
Nu este minunat să ai o problemă cu adevărat perplexă pe care să o rozi, una care generează explicații potențiale aproape nesfârșite. Ce zici de „ce caută tot acel ADN necodificator în genomuri?” – acel 98,5% din materialul genetic uman care nu produce proteine. Ca să fim corecți, descifrarea ADN-ului necodificator face pași mari prin identificarea secvențelor care sunt transcrise în ARN-uri care modulează expresia genelor, pot fi transmise transgenerațional (epigenetică) sau stabilesc programul de expresie genetică al unei celule stem sau al unei celule tisulare specifice. Cantități masive de secvențe repetate (rămășițe ale retrovirusurilor străvechi) au fost descoperite în multe genomuri și, din nou, acestea nu codifică proteine, dar cel puțin există modele credibile pentru ceea ce fac în termeni evolutivi (de la parazitism genomic la simbioză și chiar „exploatare” de către chiar genomul gazdă pentru producerea diversității genetice pe care funcționează evoluția); întâmplător, unele ADN-uri necodificatoare produc ARN-uri care reduc la tăcere aceste secvențe retrovirale și se crede că pătrunderea retrovirală în genomuri a fost presiunea selectivă pentru evoluția interferenței ARN (așa-numitul ARNi); elementele repetitive de diferite tipuri denumite și repetițiile în tandem abundă; intronii (dintre care mulți conțin tipurile de secvențe necodificatoare menționate mai sus) s-au dovedit a fi cruciali în exprimarea și reglarea genelor, în mod deosebit prin intermediul splicingului alternativ al segmentelor codificatoare pe care le separă.
Cu toate acestea, există o mulțime de probleme pe care să le ronțăiem, deoarece, deși înțelegem din ce în ce mai bine natura și originea unei mari părți a genomului necodificator și facem progrese majore în ceea ce privește „funcția” sa (definită aici ca efect avantajos, selectat evolutiv, asupra organismului gazdă), suntem departe de a explica totul și – mai mult decât atât – îl privim cu o lentilă cu mărime foarte mică, ca să spunem așa. Unul dintre lucrurile intrigante despre secvențele de ADN este faptul că o singură secvență poate „codifica” mai multe informații, în funcție de ceea ce o „citește” și în ce direcție – genomurile virale sunt exemple clasice în care genele citite într-o direcție pentru a produce o anumită proteină se suprapun cu una sau mai multe gene citite în direcția opusă (adică, din șirul complementar de ADN) pentru a produce proteine diferite. Este un pic ca și cum ai face mesaje simple cu cuvinte cu pereche inversă (un așa-numit emordnilap). De exemplu: REEDSTOPSFLOW, care, prin intermediul unui dispozitiv imaginar de citire, ar putea fi împărțit în REED STOPS FLOW. Citit invers, ar da WOLF SPOTS DEER.
Acum, dacă este un avantaj evolutiv ca două mesaje să fie codificate atât de economic – așa cum se întâmplă în cazul genomurilor virale, care tind să evolueze spre o complexitate minimă în ceea ce privește conținutul informațional, reducând astfel resursele necesare pentru reproducere – atunci mesajele însele evoluează cu un grad ridicat de constrângere. Ce înseamnă acest lucru? Ei bine, am putea formula exemplul nostru original de mesaj ca RUSH-STEM IMPEDES CURRENT, care ar încorpora aceeași informație esențială ca și REED STOPS FLOW. Cu toate acestea, acest mesaj, dacă este citit în sens invers (sau chiar în același sens, dar în bucăți diferite), nu codifică nimic suplimentar care să fie deosebit de semnificativ. Probabil că singura modalitate de a transmite simultan ambele informații din mesajele originale este chiar formularea REEDSTOPSFLOW: acesta este un sistem extrem de constrâns! Într-adevăr, dacă am studia destule exemple de fraze cu pereche inversă în limba engleză, am vedea că acestea sunt, în general, alcătuite din cuvinte destul de scurte, iar secvențelor le lipsesc anumite unități de limbă, cum ar fi articolele (the, a); dacă ne-am uita mai atent, am putea chiar detecta o reprezentare mai mare decât media a anumitor litere ale alfabetului în astfel de mesaje. Am vedea acestea ca prejudecăți în utilizarea cuvintelor și literelor care ne-ar permite, a priori, să avem o încercare de a identifica astfel de informații cu „dublă funcție”.
Acum să ne întoarcem la „litere”, „cuvinte” și „informații” codificate în genomuri. Pentru ca două informații distincte să fie codificate în aceeași bucată de secvență genetică, ne-am aștepta, în mod similar, ca constrângerile să se manifeste în prejudecăți de utilizare a cuvintelor și literelor – analogiile, respectiv, pentru secvențele de aminoacizi care constituie proteinele, și codul lor de trei litere. Prin urmare, o secvență de ADN poate codifica pentru o proteină și, în plus, pentru altceva. Acest „altceva”, potrivit lui Giorgio Bernardi, este informația care dirijează împachetarea lungimii enorme de ADN dintr-o celulă în nucleul relativ mic. În primul rând, este vorba de codul care ghidează legarea proteinelor care împachetează ADN-ul, cunoscute sub numele de histone. Bernardi se referă la acesta ca la „codul genomic” – un cod structural care definește forma și compactarea ADN-ului în forma foarte condensată cunoscută sub numele de „cromatină”.
Dar nu am început cu o explicație pentru ADN necodificator, nu pentru secvențele care codifică proteine? Da, iar în întinderile lungi de ADN necodificator vedem informații care depășesc simplele repetări, repetările în tandem și rămășițele retrovirusurilor antice: există un tip de cod la nivelul preferinței pentru perechea GC a bazelor chimice ale ADN-ului față de AT. Așa cum Bernardi trece în revistă, sintetizând lucrările sale și ale altora, inovatoare, în secvențele de bază ale genomului eucariot, conținutul de GC în unitățile organizatorice structurale ale genomului denumite „izocore” a crescut în timpul tranziției evolutive între așa-numitele organisme cu sânge rece și cele cu sânge cald. Și, în mod fascinant, această tendință a secvențelor se suprapune cu secvențe care sunt mult mai constrânse din punct de vedere funcțional: acestea sunt chiar secvențele codificatoare de proteine menționate mai devreme, iar ele – mai mult decât secvențele necodificatoare care intervin – reprezintă indiciul „codului genomic”.
Secvențele codificatoare de proteine sunt, de asemenea, împachetate și condensate în nucleu – în special atunci când nu sunt „în uz” (de ex, fiind transcrise și apoi traduse în proteine) – dar ele conțin, de asemenea, informații relativ constante privind identitățile precise ale aminoacizilor, altfel nu ar reuși să codifice corect proteinele: evoluția ar acționa asupra unor astfel de mutații într-o manieră extrem de negativă, făcând extrem de improbabil ca ele să persiste și să fie vizibile pentru noi. Dar codul de aminoacizi din ADN are o mică „capcană” care a evoluat în cele mai simple organisme unicelulare (bacterii și archaea) cu miliarde de ani în urmă: codul este parțial redundant. De exemplu, aminoacidul Treonină poate fi codificat în ADN-ul eucariot în nu mai puțin de patru moduri: ACT, ACC, ACA sau ACG. A treia literă este variabilă și, prin urmare, „disponibilă” pentru codificarea de informații suplimentare. Acest lucru este exact ceea ce se întâmplă pentru a produce „codul genomic”, în acest caz creând o prejudecată pentru formele ACC și ACG în organismele cu sânge cald. Prin urmare, constrângerea ridicată asupra acestui „cod” suplimentar – care este, de asemenea, observată în părți ale genomului care nu sunt supuse unei astfel de constrângeri ca secvențe codificatoare de proteine – este impusă de împachetarea secvențelor codificatoare de proteine care încorporează simultan două seturi de informații. Acest lucru este analog cu exemplul nostru de secvență cu dublă informație REEDSTOPSFLOW, extrem de constrânsă.
Important, totuși, constrângerea nu este la fel de strictă ca în exemplul nostru din limba engleză din cauza redundanței celei de-a treia poziții a codului de triplete pentru aminoacizi: o analogie mai bună ar fi SHE*ATE*STU*, unde asteriscul reprezintă o literă variabilă care nu face nici o diferență pentru mașina care citește componenta de trei litere a mesajului de patru litere. Am putea apoi să ne imaginăm un al doilea nivel de informații format prin adăugarea lui „D” în aceste puncte de asterisc, pentru a obține SHEDATEDSTUD (SHE DATED STUD). Imaginați-vă apoi o a doua mașină de citit care caută fraze semnificative de „natură sensibilă” care conțin o concentrație mai mare decât media de D-uri. Această mașină de citit poartă cu ea o mașină de pliere care plasează un fel de cuier la fiecare D, îndoind mesajul cu 120 de grade într-un plan. un punct în care mesajul ar trebui să fie îndoit cu 120 de grade în același plan, am ajunge la o versiune mai compactă, triunghiulară. În genomurile eucariote, polarizarea secvenței GC propusă ca fiind responsabilă de condensarea structurală se extinde în secvențele necodificatoare, dintre care unele au activități identificate, deși sunt mai puțin constrânse în secvență decât ADN-ul care codifică proteine. Acolo aceasta dirijează condensarea lor prin intermediul nucleozomilor care conțin histone pentru a forma cromatina.
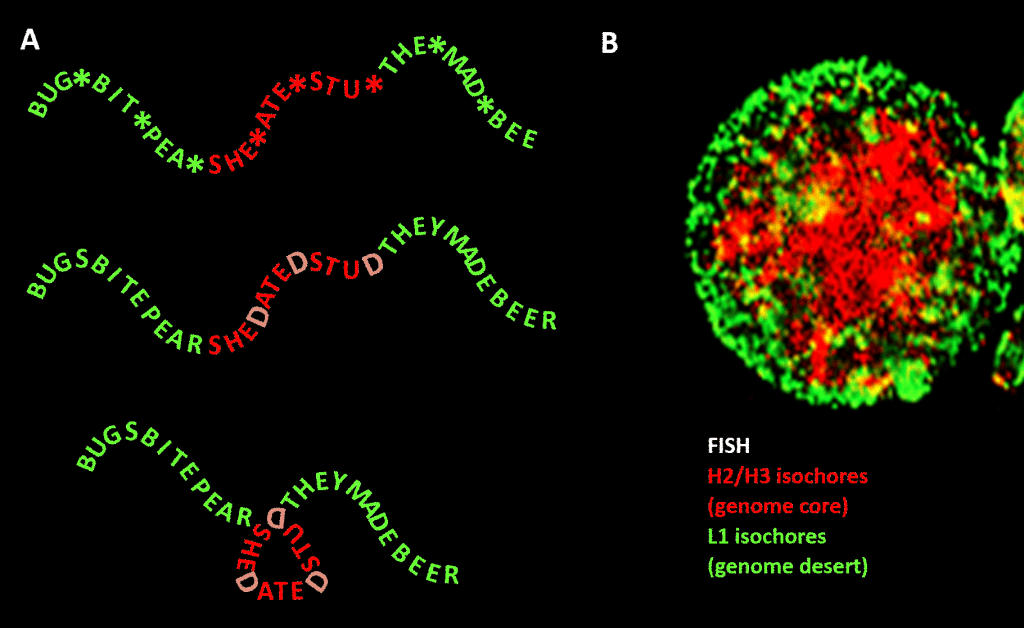
Figura. Analogia dintre condensarea unui mesaj bazat pe cuvinte și condensarea ADN-ului genomic în nucleul celular. Panoul A: Informație în informație, o secvență de cuvinte cu un al patrulea spațiu variabil care, atunci când este completat cu anumite litere, generează un alt mesaj. Un mesaj este citit de o mașină de citit trei litere; celălalt mesaj este citit de o mașină de citit care poate interpreta informațiile care se extind până la poziția a patra variabilă a secvenței. Cea de-a doua mașină de citit recunoaște informațiile „sensibile” care ar trebui ascunse și, în punctele în care apare un „D” în poziția a 4-a, pliază șirul de cuvinte, comprimând astfel partea „sensibilă” și scoțând-o din vedere. Aceasta este o analogie pentru principiul comprimării 3D genomice prin intermediul cromatinei, așa cum este descris în panoul B: o imagine de fluorescență (prin hibridizare in situ cu fluorescență – FISH) a nucleului celular. Izocorele H2/H3, al căror conținut de GC a crescut în timpul evoluției de la vertebratele cu sânge rece la cele cu sânge cald, sunt comprimate într-un nucleu de cromatină, lăsând izocorele L1 (cu un conținut mai mic de GC) la periferie într-o stare mai puțin condensată. „Codul genomic” întruchipat în traiectele cu conținut ridicat de GC ale genomului este, potrivit lui Bernardi , citit de mașinăria de poziționare a nucleozomilor din celulă și interpretat ca o secvență care trebuie să fie puternic comprimată în eucromatină. Mulțumiri: Panoul A: conceptul și realizarea figurii: Andrew Moore; Panoul B: un model FISH al izocorilor H2/H3 și L1 de la un limfocit indus de PHA – cu amabilitatea lui S. Saccone – așa cum este reprodus în Ref.].
Aceste regiuni de ADN pot fi considerate atunci ca fiind elemente importante din punct de vedere structural în formarea formei și separării corecte a secvențelor codificatoare condensate în genom, indiferent de orice altă funcție posibilă pe care o au aceste secvențe necodificatoare: în esență, aceasta ar fi o „explicație” pentru persistența în genomuri a unor secvențe cărora nu li se poate atribui nicio „funcție” (în termeni de activitate selectată de evoluție), sau, cel puțin, nicio funcție substanțială).
O ultimă analogie – de data aceasta mult mai strâns legată – ar putea fi chiar secvențele de aminoacizi din proteinele mari, care fac o varietate de răsuciri, întoarceri, pliuri etc. Ne putem minuna în fața unor astfel de structuri complicate și să ne întrebăm „dar au nevoie să fie chiar atât de complicate pentru funcția lor?”. Ei bine, poate că au nevoie pentru a condensa și poziționa părți ale proteinei în orientarea și locul exact care generează structura tridimensională care a fost selectată cu succes de evoluție. Dar, știind că „codul genomic” se suprapune peste secvențele de codificare a proteinelor, am putea chiar începe să devenim suspicioși că există și o altă presiune selectivă la lucru…
Andrew Moore, Ph.D.
Editor-șef, BioEssays