Det borde inte förvåna oss att även i delar av arvsmassan där vi inte uppenbarligen ser en ”funktionell” kod (dvs. en kod som har fastställts evolutionärt som ett resultat av någon selektiv fördel), finns det en typ av kod, men inte som något som vi tidigare har betraktat som sådan. Och tänk om den skulle göra något i tre dimensioner utöver ATGC-kodens en dimension? En artikel som just publicerats i BioEssays utforskar denna lockande möjlighet…
Är det inte underbart att ha ett riktigt förbryllande problem att gnaga på, ett problem som genererar nästan oändliga potentiella förklaringar. Vad sägs om ”vad gör allt icke-kodande DNA i genomen?” – de 98,5 procent av människans genetiska material som inte producerar proteiner. För att vara rättvis, så gör avkodningen av icke-kodande DNA stora framsteg genom identifiering av sekvenser som transkriberas till RNA som modulerar genuttrycket, kan föras vidare transgenerationellt (epigenetik) eller fastställer genuttrycksprogrammet för en stamcell eller en specifik vävnadscell. Stora mängder upprepade sekvenser (rester av gamla retrovirus) har hittats i många genom, och återigen kodar dessa inte för protein, men det finns åtminstone trovärdiga modeller för vad de gör i evolutionära termer (allt från genomisk parasitism till symbios och till och med ”utnyttjande” av själva värdgenomet för att producera den genetiska mångfald som evolutionen arbetar på); För övrigt producerar en del icke-kodande DNA RNA som tystar dessa retrovirala sekvenser, och retroviralt inträngande i genomer tros ha utgjort det selektiva trycket för evolutionen av RNA-interferens (s.k. RNAi). Repetitiva element av olika namngivna typer och tandemrepetitioner finns i överflöd. Intronerna (av vilka många innehåller ovannämnda typer av icke-kodande sekvenser) har visat sig vara avgörande för genuttryck och -reglering, framför allt genom alternativ splicing av de kodande segmenten som de avskiljer.
Det finns fortfarande gott om problem att gnaga på, för även om vi i allt högre grad förstår arten och ursprunget av en stor del av det icke-kodande genomet och gör stora framsteg när det gäller dess ”funktion” (definierad här som evolutionärt utvald, fördelaktig effekt på värdorganismen), så är vi långt ifrån att förklara allting, och – för att vara ännu mer exakt – vi betraktar det med en mycket lågt förstorande lins, så att säga. En av de fascinerande sakerna med DNA-sekvenser är att en enda sekvens kan ”koda” mer än en information beroende på vad som ”läser” den och i vilken riktning – virusgenom är klassiska exempel där gener som läses i en riktning för att producera ett visst protein överlappar med en eller flera gener som läses i motsatt riktning (dvs. från den komplementära DNA-strängen) för att producera olika proteiner. Det är lite som att göra enkla meddelanden med omvänt parade ord (en så kallad emordnilap). Till exempel: REEDSTOPSFLOW, som med hjälp av en tänkt läsanordning skulle kunna delas upp i REED STOPS FLOW. Läst baklänges skulle det ge WOLF SPOTS DEER.
Nu, om det är en evolutionär fördel för två meddelanden att kodas så ekonomiskt – vilket är fallet i virusgenom, som tenderar att utvecklas mot minsta möjliga komplexitet när det gäller informationsinnehållet, vilket minskar de nödvändiga resurserna för reproduktion – så utvecklas meddelandena i sig själva med en hög grad av begränsning. Vad innebär detta? Vi skulle kunna formulera vårt ursprungliga exempelmeddelande som RUSH-STEM IMPEDES CURRENT, vilket skulle innehålla samma viktiga information som REED STOPPAR FLOW. Men om det meddelandet läses omvänt (eller till och med i samma mening, men i olika delar) kodar det inte för något ytterligare som är särskilt meningsfullt. Det enda sättet att förmedla båda delarna av informationen i de ursprungliga meddelandena samtidigt är förmodligen själva formuleringen REEDSTOPSFLOW: det är ett mycket begränsat system! Om vi studerade tillräckligt många exempel på omvända parfraser på engelska skulle vi faktiskt se att de på det hela taget består av ganska korta ord, och att sekvenserna saknar vissa språkenheter som t.ex. artiklar (the, a); om vi tittade närmare efter skulle vi till och med kunna upptäcka att vissa bokstäver i alfabetet är mer representerade än genomsnittet i sådana meddelanden. Vi skulle se detta som en förskjutning i användningen av ord och bokstäver som på förhand skulle göra det möjligt för oss att göra ett försök att identifiera sådana ”dubbelfunktionella” delar av informationen.
Nu ska vi återgå till ”bokstäverna”, ”orden” och ”informationen” som är kodade i genomerna. Om två olika delar av informationen skulle kodas i samma bit av en genetisk sekvens skulle vi på samma sätt förvänta oss att begränsningarna skulle visa sig i form av en snedvridning av ord- och bokstavsanvändningen, vilket är analogt med aminosyresekvenser som utgör proteiner och deras trelängdsbokstavskod. En DNA-sekvens kan alltså koda för ett protein och dessutom för något annat. Detta ”något annat” är enligt Giorgio Bernardi information som styr paketeringen av den enorma DNA-längden i en cell till den relativt lilla cellkärnan. Det är i första hand koden som styr bindningen av de DNA-förpackningsproteiner som kallas histoner. Bernardi kallar detta för den ”genomiska koden” – en strukturell kod som definierar formen och komprimeringen av DNA till den starkt kondenserade form som kallas ”kromatin”.
Men började vi inte med en förklaring till icke-kodande DNA, inte till proteinkodande sekvenser? Jo, och i de långa sträckorna av icke-kodande DNA ser vi information som går utöver rena upprepningar, tandemrepetitioner och rester av gamla retrovirus: det finns en typ av kod på nivån av preferens för GC-paret av kemiska DNA-baser jämfört med AT. Som Bernardi granskar, genom att sammanfatta sitt och andras banbrytande arbete, ökade GC-innehållet i de centrala sekvenserna av det eukaryotiska genomet i de strukturella organisatoriska enheterna i genomet som benämns ”isokores” under den evolutionära övergången mellan så kallade kallblodiga och varmblodiga organismer. Och fascinerande nog överlappar denna sekvensbias med sekvenser som är mycket mer begränsade i sin funktion: dessa är just de proteinkodande sekvenser som nämndes tidigare, och de – mer än de mellanliggande icke-kodande sekvenserna – är ledtråden till den ”genomiska koden”.
Proteinkodande sekvenser är också packade och kondenserade i kärnan – särskilt när de inte är ”i bruk” (dvs, De innehåller också relativt konstant information om exakta aminosyraidentiteter, annars skulle de misslyckas med att koda proteiner korrekt: evolutionen skulle agera på sådana mutationer på ett mycket negativt sätt, vilket skulle göra det ytterst osannolikt att de skulle bestå och vara synliga för oss. Aminosyrakoden i DNA har dock en liten ”hake” som utvecklades i de enklaste encelliga organismerna (bakterier och arkéer) för miljarder år sedan: koden är delvis överflödig. Till exempel kan aminosyran tretonin kodas i eukaryotiskt DNA på inte mindre än fyra sätt: ACT, ACC, ACA eller ACG. Den tredje bokstaven är variabel och därmed ”tillgänglig” för kodning av extra information. Detta är exakt vad som händer för att skapa den ”genomiska koden”, som i det här fallet skapar en förkärlek för ACC- och ACG-formerna i varmblodiga organismer. Den höga begränsningen av denna extra ”kod” – som också ses i delar av genomet som inte är under sådan begränsning som proteinkodande sekvenser – påförs alltså av paketeringen av proteinkodande sekvenser som förkroppsligar två uppsättningar information samtidigt. Detta är analogt med vårt exempel på den starkt begränsade sekvensen med dubbel information REEDSTOPSFLOW.
Väsentligt är dock att begränsningen inte är lika strikt som i vårt exempel på det engelska språket på grund av redundansen i den tredje positionen i triplettkoden för aminosyror: en bättre analogi skulle vara SHE*ATE*STU* där asterisken står för en variabel bokstav som inte gör någon skillnad för maskinen som läser den tre bokstavsbundna komponenten av det fyrbokstaviga meddelandet. Man skulle sedan kunna tänka sig en andra informationsnivå som bildas genom att lägga till ”D” vid dessa asteriskpunkter, så att man får SHEDATEDSTUD (SHE DATED STUD). Föreställ er sedan en andra läsmaskin som letar efter meningsfulla fraser av ”känslig karaktär” som innehåller en högre koncentration av D:n än genomsnittet. Denna läsmaskin har med sig en vikningsmaskin som placerar ett slags pinne vid varje D, vilket knycker budskapet med 120 grader i ett plan. en punkt där budskapet ska böjas med 120 grader i samma plan, skulle vi få en mer kompakt, triangulär, version. I eukaryota genomer sträcker sig den GC-sekvensbias som föreslås vara ansvarig för strukturell kondensering in i icke-kodande sekvenser, av vilka vissa har identifierade aktiviteter, även om de är mindre begränsade i sekvens än proteinkodande DNA. Där styr den deras kondensering via histoninnehållande nukleosomer för att bilda kromatin.
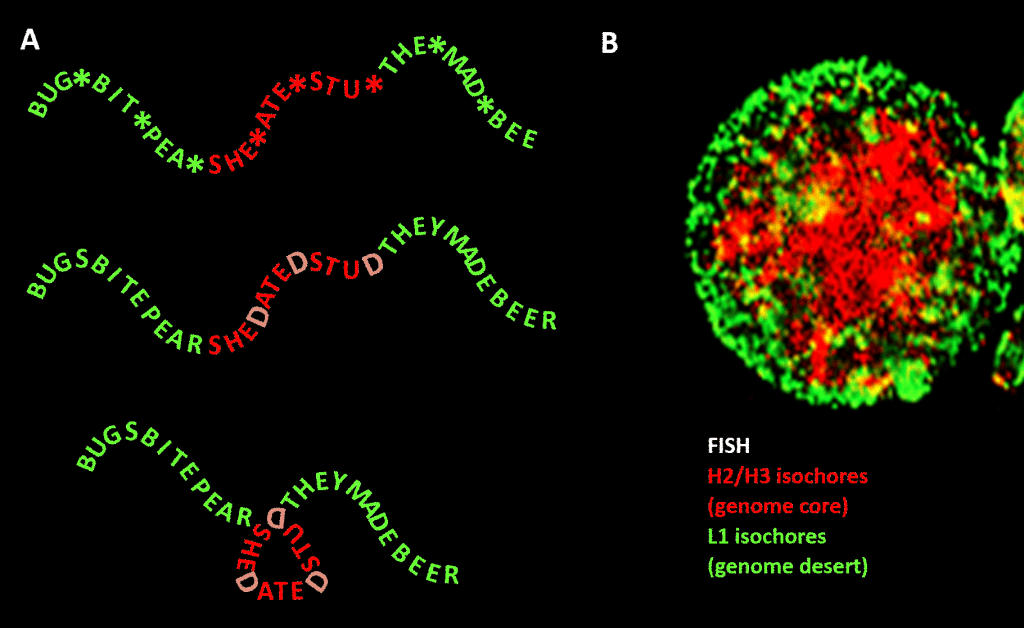
Figur. Analogi mellan kondensering av ett ordbaserat meddelande och kondensering av genomiskt DNA i cellkärnan. Panel A: Information inom information, en sekvens av ord med ett variabelt fjärde utrymme som, när det fylls med vissa bokstäver, genererar ytterligare ett meddelande. Det ena meddelandet läses av en läsmaskin med tre bokstäver, det andra av en läsmaskin som kan tolka information som sträcker sig till den fjärde variabla positionen i sekvensen. Den andra läsaren känner igen ”känslig” information som bör döljas, och på de ställen där ett ”D” förekommer i den fjärde positionen viker den ihop ordraden, vilket komprimerar den ”känsliga” delen och gör att den inte syns. Detta är en analogi till principen för genomisk 3D-komprimering via kromatin, som visas i panel B: en fluorescensbild (via Fluorescence In-Situ Hybridization – FISH) av cellkärnan. H2/H3-isokorerna, som ökade i GC-innehåll under evolutionen från kallblodiga till varmblodiga ryggradsdjur, komprimeras till en kromatinkärna och lämnar L1-isokorerna (med lägre GC-innehåll) i periferin i ett mindre komprimerat tillstånd. Den ”genomiska koden” som ingår i de höga GC-sträckorna i arvsmassan läses enligt Bernardi av cellens nukleosomplaceringsmaskineri och tolkas som en sekvens som skall komprimeras kraftigt i euchromatin. Tack: Panel A: koncept och produktion av figurer: Panel B: Ett FISH-mönster av H2/H3- och L1-isokorer från en lymfocyt som inducerats av PHA – med tillstånd av S. Saccone – som återges i Ref.]
Dessa DNA-regioner kan då betraktas som strukturellt viktiga element för att bilda den korrekta formen och separationen av kondenserade kodande sekvenser i genomet, oavsett vilken annan tänkbar funktion som dessa icke-kodande sekvenser har: i huvudsak skulle detta vara en ”förklaring” till att sekvenser som inte kan tillskrivas någon ”funktion” (i termer av evolutionärt utvald aktivitet), eller åtminstone inte någon väsentlig funktion, finns kvar i genomer.
En sista analogi – denna gång mycket närmare besläktad – skulle kunna vara själva aminosyrasekvenserna i stora proteiner, som gör en mängd vridningar, vändningar, veckningar osv. Vi kan förundras över sådana komplicerade strukturer och fråga oss ”men behöver de vara så komplicerade för sin funktion?”. Kanske måste de det för att kondensera och placera delar av proteinet i den exakta riktning och på den exakta plats som genererar den tredimensionella struktur som framgångsrikt har valts ut av evolutionen. Men med en kunskap om att den ”genomiska koden” överlappar proteinkodande sekvenser kan vi till och med börja misstänka att det finns ett annat selektivt tryck som också är verksamt…
Andrew Moore, Ph.D.
Chefredaktör, BioEssays