- Descrivere la struttura e la funzione delle proteine
- Obiettivi di apprendimento
- Aminoacidi
- Domanda pratica
- Il significato evolutivo del citocromo c
- Struttura della proteina
- Struttura primaria
- Struttura secondaria
- Struttura terziaria
- Struttura quaternaria
- Denaturazione e ripiegamento delle proteine
- Funzione delle proteine
- In sintesi: Le proteine
- Verifica la tua comprensione
Descrivere la struttura e la funzione delle proteine
Le proteine sono polimeri di amminoacidi. Ogni amminoacido contiene un carbonio centrale, un idrogeno, un gruppo carbossilico, un gruppo amminico e un gruppo R variabile. Il gruppo R specifica a quale classe di aminoacidi appartiene: catene laterali idrofile elettricamente cariche, catene laterali polari ma non cariche, catene laterali idrofobiche non polari e casi speciali.
Le proteine hanno diversi “strati” di struttura: primaria, secondaria, terziaria, quaternaria.
Le proteine hanno una varietà di funzioni nelle cellule. Le funzioni principali includono la funzione di enzimi, recettori, molecole di trasporto, proteine regolatrici dell’espressione genica, e così via. Gli enzimi sono catalizzatori biologici che accelerano una reazione chimica senza essere alterati in modo permanente. Hanno “siti attivi” dove si lega il substrato/reagente, e possono essere attivati o inibiti (inibitori competitivi e/o non competitivi).
Obiettivi di apprendimento
- Dimostrare familiarità con le unità monomeriche delle proteine: aminoacidi
- Definire i diversi strati della struttura delle proteine
- Identificare diverse funzioni principali delle proteine
Aminoacidi
Le proteine sono una delle molecole organiche più abbondanti nei sistemi viventi e hanno la gamma più varia di funzioni di tutte le macromolecole. Le proteine possono essere strutturali, regolatrici, contrattili o protettive; possono servire per il trasporto, lo stoccaggio o le membrane; o possono essere tossine o enzimi. Ogni cellula di un sistema vivente può contenere migliaia di proteine, ognuna con una funzione unica. Le loro strutture, come le loro funzioni, variano molto. Sono tutte, comunque, polimeri di aminoacidi, disposti in una sequenza lineare.

Figura 1. Gli amminoacidi hanno un carbonio centrale asimmetrico a cui sono attaccati un gruppo amminico, un gruppo carbossilico, un atomo di idrogeno e una catena laterale (gruppo R).
Gli amminoacidi sono i monomeri che costituiscono le proteine. Ogni amminoacido ha la stessa struttura fondamentale, che consiste in un atomo di carbonio centrale, noto anche come carbonio alfa (α), legato a un gruppo amminico (NH2), un gruppo carbossilico (COOH) e a un atomo di idrogeno. Ogni aminoacido ha anche un altro atomo o gruppo di atomi legati all’atomo centrale conosciuto come gruppo R (Figura 1).
Il nome “aminoacido” deriva dal fatto che contengono sia il gruppo amminico che il gruppo carbossilico-acido nella loro struttura di base. Come detto, ci sono 20 aminoacidi presenti nelle proteine. Dieci di questi sono considerati aminoacidi essenziali nell’uomo perché il corpo umano non può produrli e sono ottenuti dalla dieta.
Per ogni aminoacido, il gruppo R (o catena laterale) è diverso (Figura 2).
Domanda pratica

Figura 2. Ci sono 20 amminoacidi comuni che si trovano comunemente nelle proteine, ognuno con un diverso gruppo R (gruppo variante) che determina la sua natura chimica.
Quali categorie di amminoacidi ti aspetteresti di trovare sulla superficie di una proteina solubile, e quali ti aspetteresti di trovare all’interno? Quale distribuzione di amminoacidi ti aspetteresti di trovare in una proteina incorporata in un bilayer lipidico?
La natura chimica della catena laterale determina la natura dell’aminoacido (cioè, se è acido, basico, polare o non polare). Per esempio, l’aminoacido glicina ha un atomo di idrogeno come gruppo R. Aminoacidi come la valina, la metionina e l’alanina sono di natura non polare o idrofoba, mentre aminoacidi come la serina, la treonina e la cisteina sono polari e hanno catene laterali idrofile. Le catene laterali di lisina e arginina sono caricate positivamente, e quindi questi aminoacidi sono anche conosciuti come aminoacidi basici. La prolina ha un gruppo R che è legato al gruppo amminico, formando una struttura ad anello. La prolina è un’eccezione alla struttura standard di un aminoacido poiché il suo gruppo amminico non è separato dalla catena laterale (Figura 2).
Gli aminoacidi sono rappresentati da una singola lettera maiuscola o da un’abbreviazione di tre lettere. Per esempio, la valina è conosciuta con la lettera V o con il simbolo a tre lettere val. Proprio come alcuni acidi grassi sono essenziali per una dieta, anche alcuni aminoacidi sono necessari. Sono conosciuti come aminoacidi essenziali, e nell’uomo includono isoleucina, leucina e cisteina. Gli aminoacidi essenziali si riferiscono a quelli necessari per la costruzione di proteine nel corpo, anche se non prodotti dal corpo; quali aminoacidi sono essenziali varia da organismo a organismo.

Figura 3. La formazione del legame peptidico è una reazione di sintesi di disidratazione. Il gruppo carbossilico di un aminoacido è legato al gruppo amminico dell’aminoacido in arrivo. Nel processo, viene rilasciata una molecola d’acqua.
La sequenza e il numero di amminoacidi determinano alla fine la forma, la dimensione e la funzione della proteina. Ogni amminoacido è attaccato ad un altro amminoacido da un legame covalente, noto come legame peptidico, che si forma da una reazione di disidratazione. Il gruppo carbossilico di un aminoacido e il gruppo amminico dell’aminoacido in arrivo si combinano, rilasciando una molecola d’acqua. Il legame risultante è il legame peptidico (Figura 3).
I prodotti formati da tali legami sono chiamati peptidi. Quando più aminoacidi si uniscono a questa catena crescente, la catena risultante è nota come polipeptide. Ogni polipeptide ha un gruppo amminico libero ad un’estremità. Questa estremità è chiamata terminale N, o terminale amminico, e l’altra estremità ha un gruppo carbossilico libero, noto anche come terminale C o carbossilico. Mentre i termini polipeptide e proteina sono talvolta usati in modo intercambiabile, un polipeptide è tecnicamente un polimero di amminoacidi, mentre il termine proteina è usato per un polipeptide o polipeptidi che si sono combinati insieme, spesso hanno legato gruppi prostetici non peptidici, hanno una forma distinta, e hanno una funzione unica. Dopo la sintesi proteica (traduzione), la maggior parte delle proteine vengono modificate. Queste sono conosciute come modifiche post-traslazionali. Possono essere sottoposte a scissione, fosforilazione o possono richiedere l’aggiunta di altri gruppi chimici. Solo dopo queste modifiche la proteina è completamente funzionale.
Il significato evolutivo del citocromo c
Il citocromo c è un componente importante della catena di trasporto degli elettroni, una parte della respirazione cellulare, e si trova normalmente nell’organello cellulare, il mitocondrio. Questa proteina ha un gruppo prostetico eme, e lo ione centrale dell’eme viene alternativamente ridotto e ossidato durante il trasferimento di elettroni. Poiché il ruolo di questa proteina essenziale nella produzione di energia cellulare è cruciale, è cambiata molto poco nel corso di milioni di anni. Il sequenziamento delle proteine ha dimostrato che c’è una quantità considerevole di omologia nella sequenza degli aminoacidi del citocromo c tra le diverse specie; in altre parole, la parentela evolutiva può essere valutata misurando le somiglianze o le differenze tra le sequenze di DNA o proteine di varie specie.
Gli scienziati hanno determinato che il citocromo c umano contiene 104 aminoacidi. Per ogni molecola di citocromo c di diversi organismi che è stata sequenziata fino ad oggi, 37 di questi aminoacidi appaiono nella stessa posizione in tutti i campioni di citocromo c. Questo indica che potrebbe esserci stato un antenato comune. Confrontando le sequenze della proteina umana e dello scimpanzé, non è stata trovata alcuna differenza di sequenza. Quando le sequenze umane e di scimmia rhesus sono state confrontate, la singola differenza trovata era in un aminoacido. In un altro confronto, il sequenziamento da umano a lievito mostra una differenza nella 44a posizione.
Struttura della proteina
Come discusso in precedenza, la forma di una proteina è fondamentale per la sua funzione. Per esempio, un enzima può legarsi a un substrato specifico in un sito noto come sito attivo. Se questo sito attivo è alterato a causa di cambiamenti locali o cambiamenti nella struttura generale della proteina, l’enzima potrebbe non essere in grado di legarsi al substrato. Per capire come la proteina ottiene la sua forma finale o conformazione, dobbiamo capire i quattro livelli della struttura proteica: primario, secondario, terziario e quaternario.
Struttura primaria
La sequenza unica di aminoacidi in una catena polipeptidica è la sua struttura primaria. Per esempio, l’ormone pancreatico insulina ha due catene polipeptidiche, A e B, e sono collegate tra loro da legami disolfuro. L’aminoacido terminale N della catena A è la glicina, mentre l’aminoacido terminale C è l’asparagina (Figura 4). Le sequenze di aminoacidi nelle catene A e B sono uniche per l’insulina.
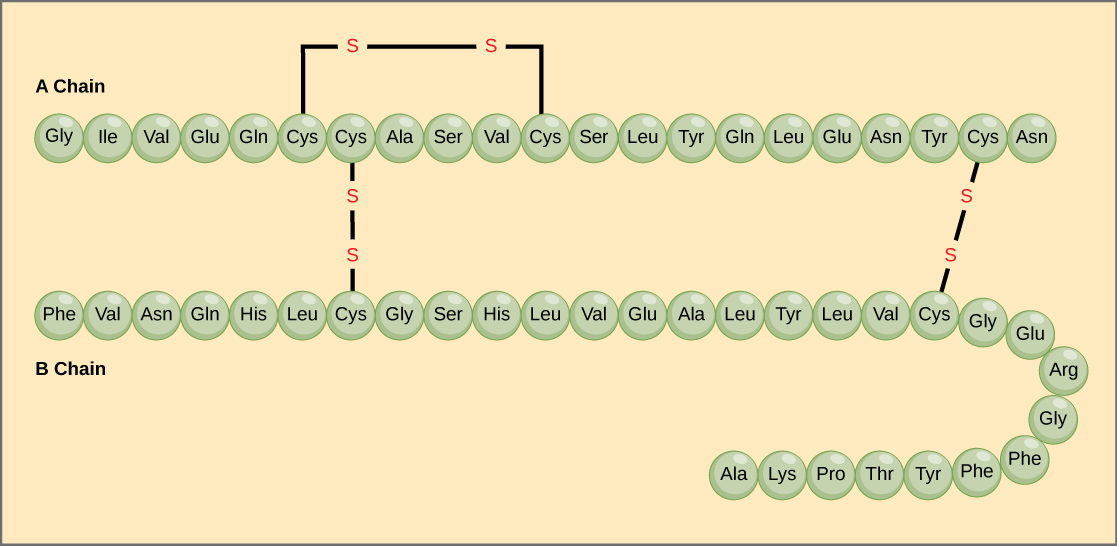
Figura 4. L’insulina del siero bovino è un ormone proteico composto da due catene peptidiche, A (lunga 21 aminoacidi) e B (lunga 30 aminoacidi). In ogni catena, la struttura primaria è indicata da abbreviazioni di tre lettere che rappresentano i nomi degli aminoacidi nell’ordine in cui sono presenti. L’aminoacido cisteina (cys) ha un gruppo sulfidrilico (SH) come catena laterale. Due gruppi sulfidrilici possono reagire in presenza di ossigeno per formare un legame disolfuro (S-S). Due legami disolfuro collegano insieme le catene A e B, e un terzo aiuta la catena A a piegarsi nella forma corretta. Si noti che tutti i legami disolfuro sono della stessa lunghezza, ma sono disegnati di dimensioni diverse per chiarezza.
La sequenza unica per ogni proteina è determinata in ultima analisi dal gene che codifica la proteina. Un cambiamento nella sequenza nucleotidica della regione codificante del gene può portare all’aggiunta di un diverso amminoacido alla catena polipeptidica in crescita, causando un cambiamento nella struttura e nella funzione della proteina. Nell’anemia falciforme, la catena β dell’emoglobina (una piccola porzione della quale è mostrata nella Figura 5) ha una singola sostituzione aminoacidica, che causa un cambiamento nella struttura e nella funzione della proteina.
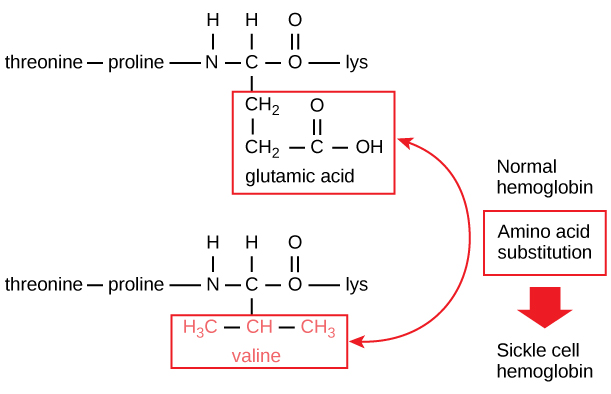
Figura 5. La catena beta dell’emoglobina è lunga 147 residui, eppure una singola sostituzione aminoacidica porta all’anemia falciforme. Nell’emoglobina normale, l’aminoacido in posizione sette è il glutammato. Nell’emoglobina falciforme, questo glutammato è sostituito da una valina.
In particolare, l’aminoacido acido glutammico è sostituito dalla valina nella catena β. La cosa più notevole da considerare è che una molecola di emoglobina è composta da due catene alfa e due catene beta che consistono ciascuna di circa 150 aminoacidi. La molecola, quindi, ha circa 600 aminoacidi. La differenza strutturale tra una molecola di emoglobina normale e una molecola di falcemia – che diminuisce drammaticamente l’aspettativa di vita – è un singolo aminoacido dei 600. Ciò che è ancora più notevole è che quei 600 aminoacidi sono codificati da tre nucleotidi ciascuno, e la mutazione è causata da un singolo cambiamento di base (mutazione puntiforme), 1 su 1800 basi.
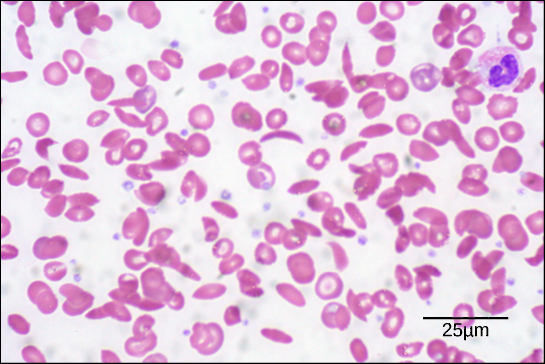
Figura 6. In questo striscio di sangue, visualizzato con un ingrandimento di 535x usando la microscopia a campo chiaro, le cellule falciformi sono a forma di mezzaluna, mentre le cellule normali sono a forma di disco. (credito: modifica del lavoro di Ed Uthman; dati in scala da Matt Russell)
A causa di questo cambiamento di un aminoacido nella catena, le molecole di emoglobina formano lunghe fibre che distorcono i globuli rossi biconcavi, o a forma di disco, e assumono una forma a mezzaluna o “falce”, che intasa le arterie (Figura 6). Questo può portare a una miriade di gravi problemi di salute come dispnea, vertigini, mal di testa e dolori addominali per le persone colpite da questa malattia.
Struttura secondaria
Il ripiegamento locale del polipeptide in alcune regioni dà origine alla struttura secondaria della proteina. Le più comuni sono le strutture ad α-elica e a foglio β-pleato (Figura 7). Entrambe le strutture sono la struttura ad α-elica – l’elica tenuta in forma da legami idrogeno. I legami a idrogeno si formano tra l’atomo di ossigeno nel gruppo carbonilico di un aminoacido e un altro aminoacido che si trova quattro aminoacidi più avanti nella catena.
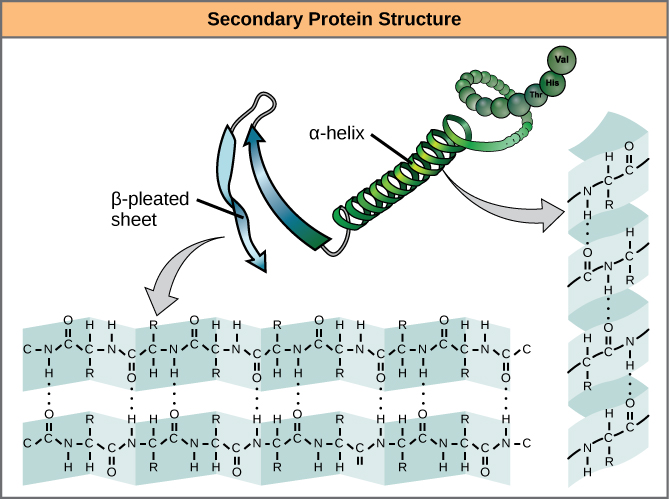
Figura 7. L’α-elica e il foglio β-pleato sono strutture secondarie delle proteine che si formano a causa del legame a idrogeno tra i gruppi carbonilici e amminici nella spina dorsale del peptide. Alcuni amminoacidi hanno una propensione a formare un’α-elica, mentre altri hanno una propensione a formare un foglio β-pleato.
Ogni giro elicoidale in un’alfa-elica ha 3,6 residui di amminoacidi. I gruppi R (i gruppi varianti) del polipeptide sporgono dalla catena dell’α-elica. Nel foglio a β-elica, le “pieghe” sono formate dal legame idrogeno tra gli atomi sulla spina dorsale della catena polipeptidica. I gruppi R sono attaccati ai carboni e si estendono sopra e sotto le pieghe della piega. I segmenti pieghettati si allineano paralleli o antiparalleli l’uno all’altro, e i legami a idrogeno si formano tra l’atomo di azoto parzialmente positivo del gruppo amminico e l’atomo di ossigeno parzialmente negativo del gruppo carbonilico della spina dorsale del peptide. Le strutture α-elica e foglio β-pleato si trovano nella maggior parte delle proteine globulari e fibrose e svolgono un importante ruolo strutturale.
Struttura terziaria
L’unica struttura tridimensionale di un polipeptide è la sua struttura terziaria (Figura 8). Questa struttura è in parte dovuta alle interazioni chimiche che agiscono sulla catena polipeptidica. Principalmente, le interazioni tra i gruppi R creano la complessa struttura terziaria tridimensionale di una proteina. La natura dei gruppi R che si trovano negli aminoacidi coinvolti può contrastare la formazione dei legami idrogeno descritti per le strutture secondarie standard. Per esempio, i gruppi R con cariche simili sono respinti l’uno dall’altro e quelli con cariche diverse sono attratti l’uno dall’altro (legami ionici). Quando avviene il ripiegamento delle proteine, i gruppi R idrofobici degli aminoacidi non polari si trovano all’interno della proteina, mentre i gruppi R idrofili si trovano all’esterno. I primi tipi di interazioni sono noti anche come interazioni idrofobiche. L’interazione tra le catene laterali della cisteina forma legami disolfuro in presenza di ossigeno, l’unico legame covalente che si forma durante il ripiegamento delle proteine.
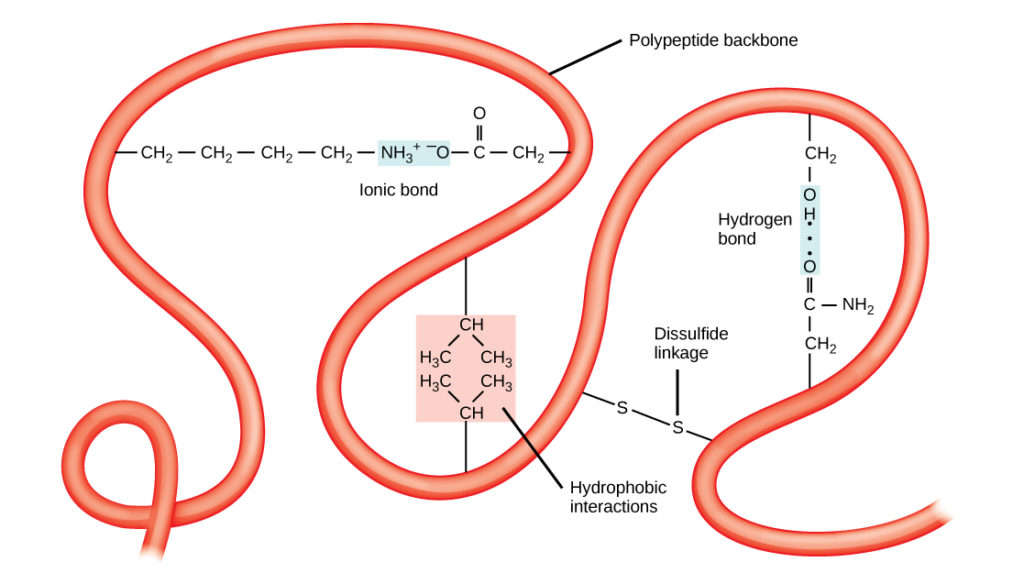
Figura 8. La struttura terziaria delle proteine è determinata da una varietà di interazioni chimiche. Queste includono interazioni idrofobiche, legame ionico, legame idrogeno e legami disolfuro.
Tutte queste interazioni, deboli e forti, determinano la forma tridimensionale finale della proteina. Quando una proteina perde la sua forma tridimensionale, può non essere più funzionale.
Struttura quaternaria
In natura, alcune proteine sono formate da diversi polipeptidi, noti anche come subunità, e l’interazione di queste subunità forma la struttura quaternaria. Le interazioni deboli tra le subunità aiutano a stabilizzare la struttura generale. Per esempio, l’insulina (una proteina globulare) ha una combinazione di legami idrogeno e legami disolfuro che la rendono per lo più agglomerata a forma di palla. L’insulina inizia come un singolo polipeptide e perde alcune sequenze interne in presenza di modifiche post-traslazionali dopo la formazione dei legami disolfuro che tengono insieme le catene rimanenti. La seta (una proteina fibrosa), invece, ha una struttura a fogli β-pleati che è il risultato del legame idrogeno tra le diverse catene.
I quattro livelli di struttura proteica (primaria, secondaria, terziaria e quaternaria) sono illustrati nella Figura 9.
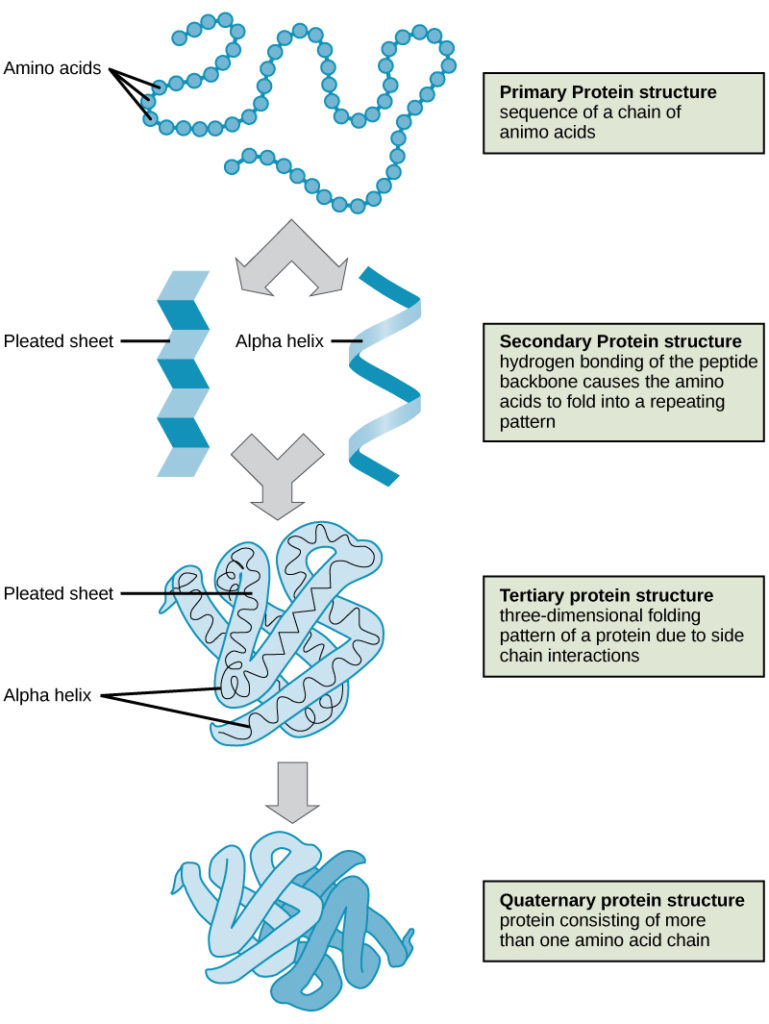
Figura 9. I quattro livelli della struttura proteica possono essere osservati in queste illustrazioni. (credito: modifica del lavoro del National Human Genome Research Institute)
Denaturazione e ripiegamento delle proteine
Ogni proteina ha la sua unica sequenza e forma che sono tenute insieme da interazioni chimiche. Se la proteina è soggetta a cambiamenti di temperatura, pH, o esposizione a sostanze chimiche, la struttura della proteina può cambiare, perdendo la sua forma senza perdere la sua sequenza primaria in ciò che è noto come denaturazione. La denaturazione è spesso reversibile perché la struttura primaria del polipeptide viene conservata nel processo se l’agente denaturante viene rimosso, permettendo alla proteina di riprendere la sua funzione. A volte la denaturazione è irreversibile, portando alla perdita di funzione. Un esempio di denaturazione irreversibile delle proteine è quando un uovo viene fritto. La proteina albumina nell’albume liquido viene denaturata quando viene messa in una padella calda. Non tutte le proteine sono denaturate ad alte temperature; per esempio, i batteri che sopravvivono nelle sorgenti calde hanno proteine che funzionano a temperature vicine all’ebollizione. Lo stomaco è anche molto acido, ha un pH basso, e denatura le proteine come parte del processo di digestione; tuttavia, gli enzimi digestivi dello stomaco mantengono la loro attività in queste condizioni.
Il ripiegamento delle proteine è fondamentale per la loro funzione. Originariamente si pensava che le proteine stesse fossero responsabili del processo di ripiegamento. Solo recentemente si è scoperto che spesso ricevono assistenza nel processo di ripiegamento da aiutanti proteici noti come chaperoni (o chaperonine) che si associano alla proteina bersaglio durante il processo di ripiegamento. Essi agiscono impedendo l’aggregazione dei polipeptidi che compongono la struttura proteica completa, e si dissociano dalla proteina una volta che la proteina bersaglio è piegata.
Funzione delle proteine
I principali tipi e funzioni delle proteine sono elencati nella Tabella 1.
| Tabella 1. Tipi di proteine e funzioni | ||
|---|---|---|
| Tipo | Esempi | Funzioni |
| Ezimi digestivi | Amilasi, lipasi, pepsina, tripsina | Aiutano nella digestione del cibo catabolizzando i nutrienti in unità monomeriche |
| Trasporto | Emoglobina, albumina | Porta sostanze nel sangue o nella linfa in tutto il corpo |
| Strutturale | Actina, tubulina, cheratina | Costruiscono diverse strutture, come il citoscheletro |
| Ormoni | Insulina, tiroxina | Coordinano l’attività di diversi sistemi corporei |
| Difesa | Immunoglobuline | Proteggono il corpo da agenti patogeni esterni |
| Contrattili | Actina, Miosina | Effetto della contrazione muscolare |
| Stoccaggio | Proteine di stoccaggio dei legumi, albume d’uovo (albumina) | Forniscono il nutrimento nel primo sviluppo dell’embrione e della piantina |
Due tipi speciali e comuni di proteine sono gli enzimi e gli ormoni. Gli enzimi, che sono prodotti dalle cellule viventi, sono catalizzatori in reazioni biochimiche (come la digestione) e sono solitamente proteine complesse o coniugate. Ogni enzima è specifico per il substrato (un reagente che si lega a un enzima) su cui agisce. L’enzima può aiutare nelle reazioni di scomposizione, riorganizzazione o sintesi. Gli enzimi che scompongono i loro substrati sono chiamati enzimi catabolici, gli enzimi che costruiscono molecole più complesse dai loro substrati sono chiamati enzimi anabolici, e gli enzimi che influenzano il tasso di reazione sono chiamati enzimi catalitici. Va notato che tutti gli enzimi aumentano il tasso di reazione e, pertanto, sono considerati catalizzatori organici. Un esempio di enzima è l’amilasi salivare, che idrolizza il suo substrato amilosio, un componente dell’amido.
Gli ormoni sono molecole di segnalazione chimica, di solito piccole proteine o steroidi, secreti da cellule endocrine che agiscono per controllare o regolare specifici processi fisiologici, tra cui la crescita, lo sviluppo, il metabolismo e la riproduzione. Per esempio, l’insulina è un ormone proteico che aiuta a regolare il livello di glucosio nel sangue.
Le proteine hanno forme e pesi molecolari diversi; alcune proteine sono di forma globosa mentre altre sono di natura fibrosa. Per esempio, l’emoglobina è una proteina globulare, ma il collagene, che si trova nella nostra pelle, è una proteina fibrosa. La forma delle proteine è fondamentale per la loro funzione, e questa forma è mantenuta da molti tipi diversi di legami chimici. I cambiamenti di temperatura, di pH e l’esposizione a sostanze chimiche possono portare a cambiamenti permanenti nella forma della proteina, con conseguente perdita di funzione, nota come denaturazione. Tutte le proteine sono costituite da diverse disposizioni degli stessi 20 tipi di aminoacidi.
In sintesi: Le proteine
Le proteine sono una classe di macromolecole che svolgono una vasta gamma di funzioni per la cellula. Aiutano il metabolismo fornendo supporto strutturale e agendo come enzimi, trasportatori o ormoni. Gli elementi costitutivi delle proteine (monomeri) sono gli aminoacidi. Ogni aminoacido ha un carbonio centrale che è legato a un gruppo amminico, un gruppo carbossilico, un atomo di idrogeno e un gruppo R o catena laterale. Ci sono 20 aminoacidi comuni, ognuno dei quali differisce nel gruppo R. Ogni amminoacido è legato ai suoi vicini da un legame peptidico. Una lunga catena di aminoacidi è nota come polipeptide.
Le proteine sono organizzate a quattro livelli: primario, secondario, terziario e (opzionale) quaternario. La struttura primaria è la sequenza unica di amminoacidi. Il ripiegamento locale del polipeptide per formare strutture come l’elica α e il foglio β-pleato costituisce la struttura secondaria. La struttura tridimensionale complessiva è la struttura terziaria. Quando due o più polipeptidi si combinano per formare la struttura proteica completa, la configurazione è nota come struttura quaternaria di una proteina. La forma delle proteine e la loro funzione sono strettamente collegate; qualsiasi cambiamento di forma causato dai cambiamenti di temperatura o di pH può portare alla denaturazione della proteina e alla perdita di funzione.
Verifica la tua comprensione
Rispondi alla domanda(e) qui sotto per vedere quanto hai capito gli argomenti trattati nella sezione precedente. Questo breve quiz non conta per il tuo voto in classe, e puoi ripeterlo un numero illimitato di volte.
Utilizza questo quiz per verificare la tua comprensione e decidere se (1) studiare ulteriormente la sezione precedente o (2) passare alla sezione successiva.